Packages R pour controler son travail et ainsi mieux optimiser, réutiliser et communiquer autour de ses analyses
Journées du PEPI IBIS 2023
September 15, 2023
Reproductibilité, pour quoi faire ?

Derrière la reproductibilité :
la transparence dans la recherche

Expliquer pour justifier et comprendre
Refaire pour vérifier, corriger et réutiliser
- Vous oblige à vérifier votre travail (partage des données + code)
- Votre futur vous-même vous remerciera
- Et vos collègues aussi
- En étant reproductible, vous renforcez votre crédibilité et votre réputation
- La reproductibilité favorise la confiance dans le processus scientifique
Vous contribuez à l’accélération des progrès scientifiques !
et vous ne perdez pas de temps …

Un des principes FAIR

Spectre de la reproctibilité

- Ne pas avoir peur d’avancer à petit pas, marche après marche
- Processus itératif et progressif
Controler ses sources (script & données)

Contrôle des versions (git)

- Enregistrer les modifications apportées à un ensemble de fichiers
- Suivre l’historique et revoir toutes les modifications
- Revenir à des versions antérieures
- Travailler collaborativement sur des fonctionnalités parallèles
Ça marche avec des scripts et des codes, des protocoles & de la documentation, des rapports, n’importe quel document !
Cycle de vie de la donnée & PGD
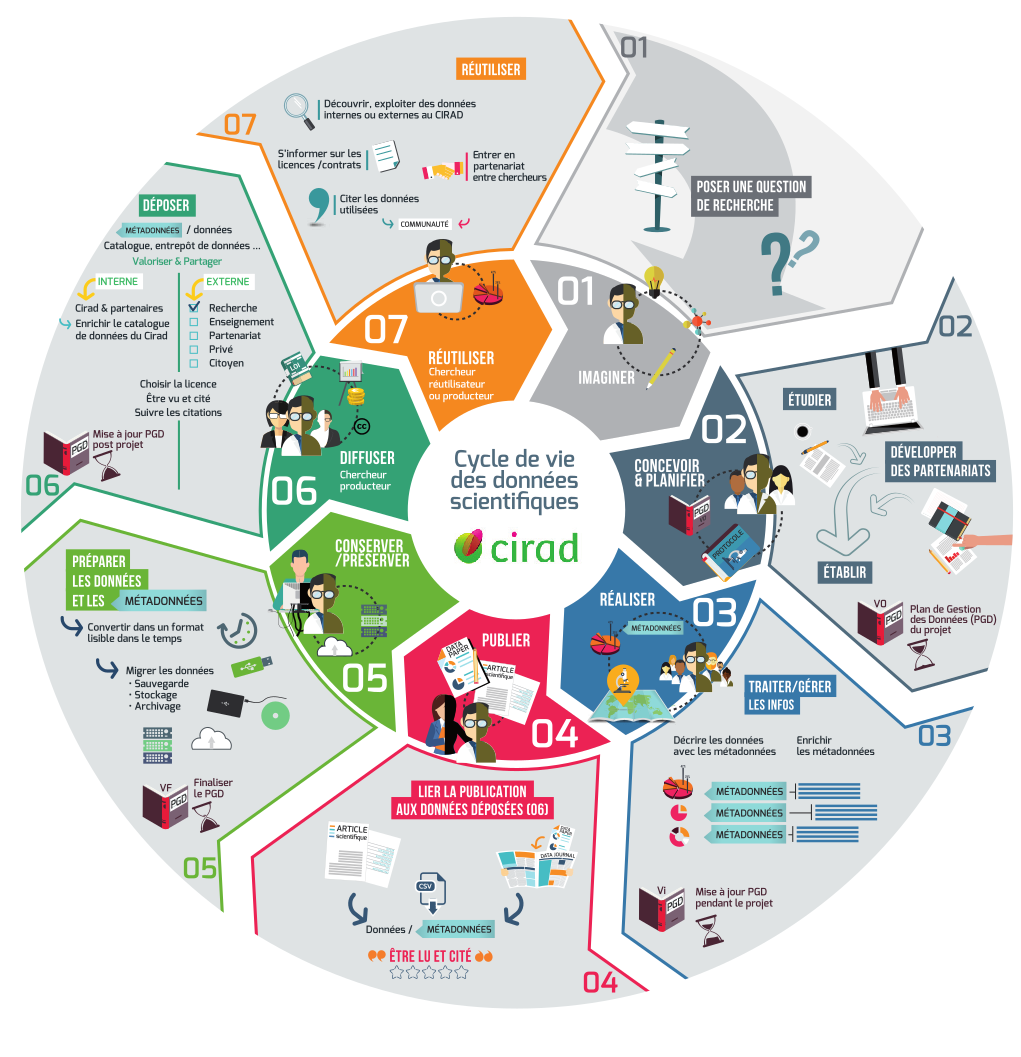
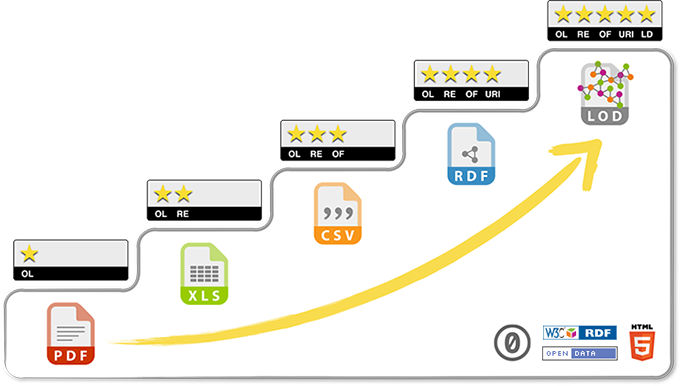
Open Data 5★
OL★ : Open LicenseRE★ : machine REadableOF★ : Open FormatURI★ : Uniform Resource IdentifierLD★ : Linked Data
Comment on fait ?
- Rédiger un Plan de Gestion de Données
- Définir un espace collaboratif unique
- nomenclature, sauvegarde, sécurité, …
- Décrire les métadonnées et un vocabulaire contrôlé
- Déposer les données dans un entrepôt international
- Contrôler son environnement, son workflow, ses documents
Ca tombe bien, c’est la suite de l’exposé !
Travailler dans un environnement controlé

Constat
While preparing a manuscript, to our surprise, attempts by team members to replicate these results produced different calculated NMR chemical shifts despite using the same Gaussian files and the same procedure outlined by Willoughby et al. […] these conclusions were based on chemical shifts that appeared to depend on the computer system on which step 15 of that protocol was performed.
[7]
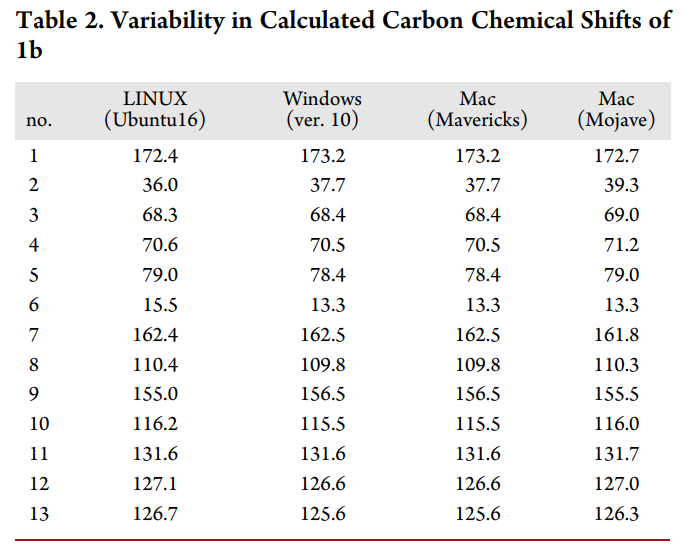
Problématique
Si un script ne fonctionne que sur l’ordi où il a été développé, il mourra avec cet ordi
Chez moi ça marche.
- Quels packages utilisés ? avec quelle version ?
- Quelle version de R ?
- Et quel OS ?
renv
renvpermet une gestion fine des versions des packages au sein de chaque projet- Pas d’effet de la mise à jour d’un package sur les autres projets
- Les versions des packages tracées dans un fichier
renv.lock - Particulièrement adapté à une utilisation avec git
renv - workflow
Initialisation d’un nouvel environnement local pour le projet avec une bibliothèque privée
Sauvegarde de l’état de la bibliothèque privée du projet. Les packages utilisés et leur version sont détaillés dans le fichier
renv.lock
Docker
Allez plus loin, partagez votre ordinateur
Dockerest un outil de conteneurisation- Un conteneur = un OS (+ du code)
- L’image créée est facilement partageable et exécutable sur un autre ordi, un autre OS
- Version allégée de la virtualisation
Docker - workflow
dockerfile
Travailler avec un workflow d’analyses controlé

Data Science
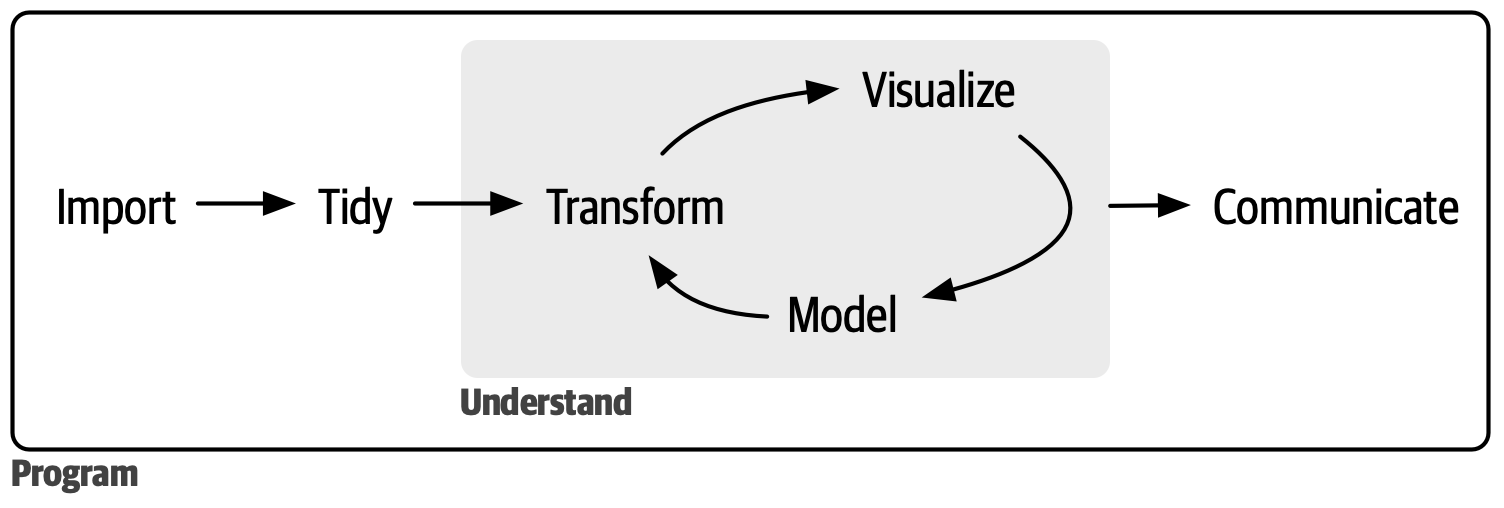
targets
Ce package permet de structurer un pipeline d’analyses sous une forme bien précise composé d’étapes écrit dans un schéma global (workflow). On pourrait le comparer à un petit snakemake ou nextflow sous R.
Facilite la parallélisation.
Philosophie
Écrire un pipeline d’analyse sous la forme d’un workflow dont chacune des étapes est reliée et dépendante les une des autres. Le but est de structurer le workflow en étapes prédéfinies et toutes structurées de la même manière (une entrée, une fonction, une sortie) et dont leur état est référencé lors de l’exécution du pipeline.
Schéma
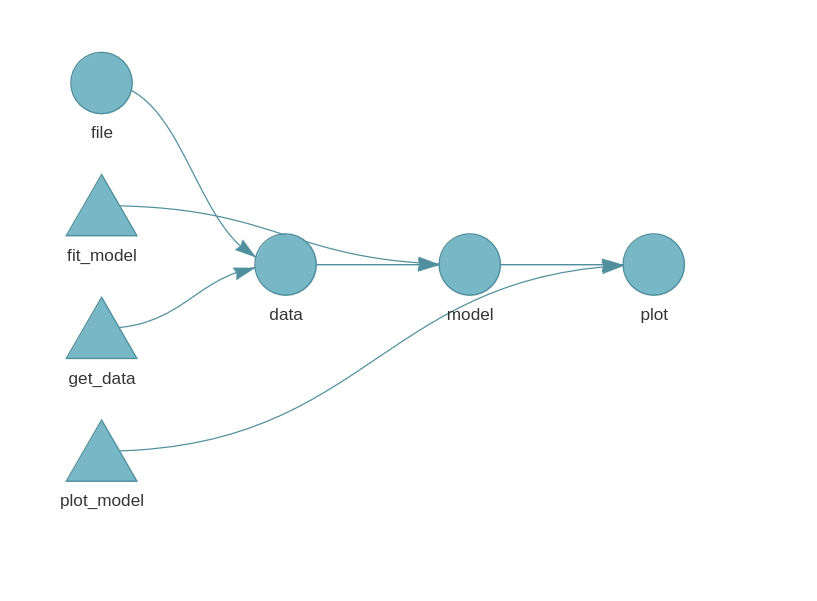
Structure du projet
├── _targets.R : le script d'execution principal
├── data/
│ ├── robject.RData : un objet R
│ ├── data.csv : les données
├── R/
│ ├── functionsMain.R : les fonctions principales utilisées pour réaliser des analyses
│ ├── functionsPlots.R : les fonctions utilisées pour réaliser les graphiques
│ ├── functionsTests.R : les fonctions secondaires
├── _output/
│ ├── output.csv : fichiers de sortie
├── _targets/
│ ├── meta/
│ ├── objects/
│ ├── user/
│ ├── workspaces/_targets.R

Execution du pipeline
La fonction tar_make() exécute le pipeline dans son ensemble en respectant l’ordre des étapes écrites dans le fichier _targets.R.
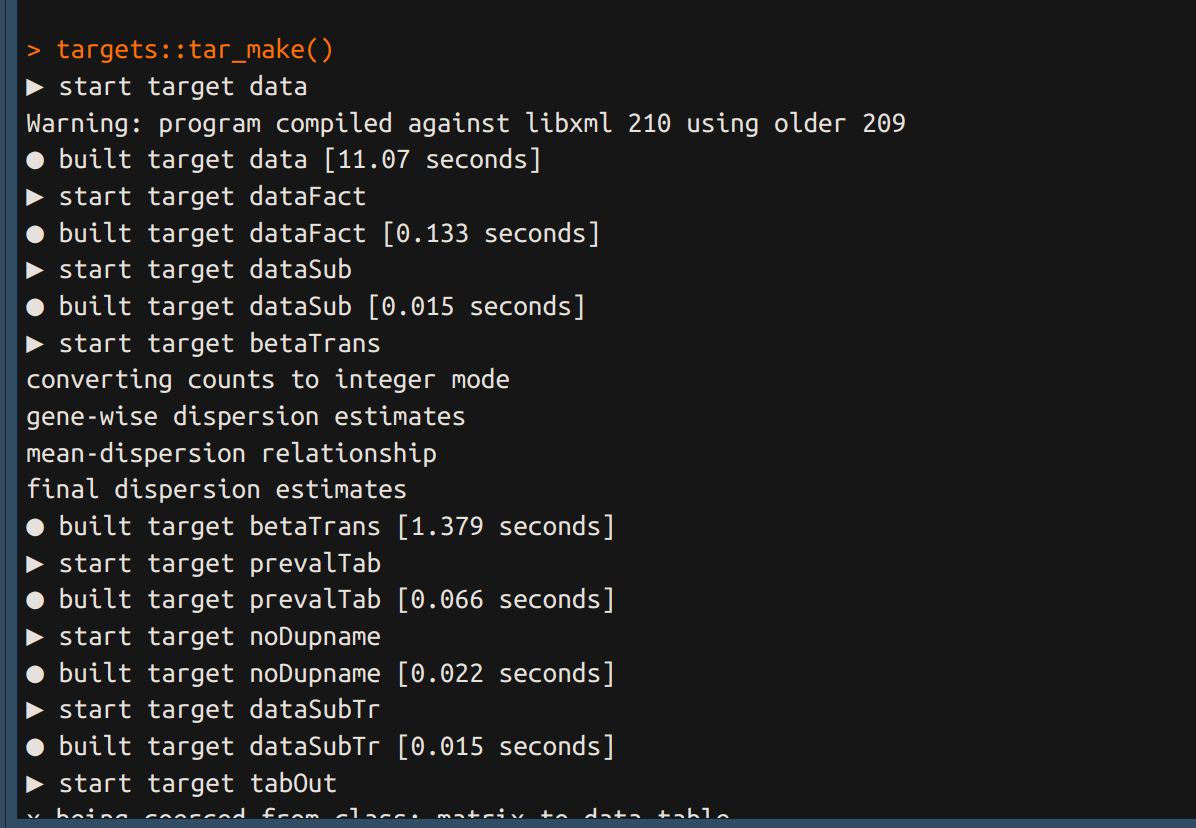
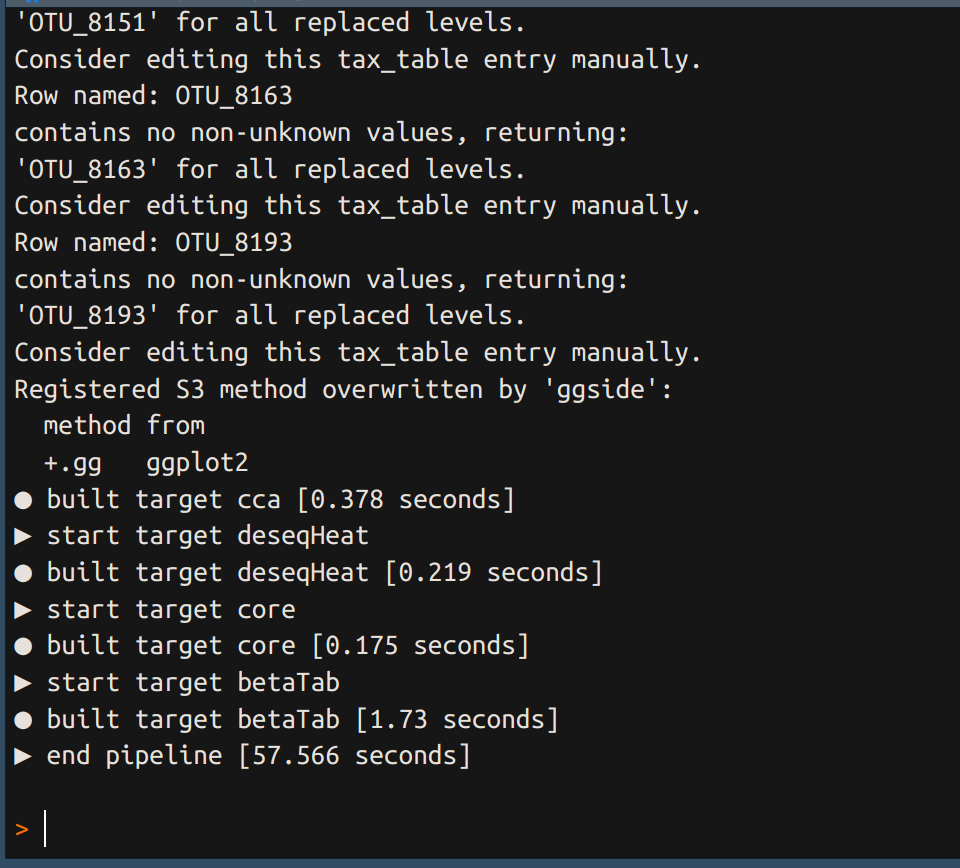
Ré-execution du pipeline
Quand on exécutera à nouveau le pipeline seules les étapes ayant été modifiées seront de nouveau exécutées.
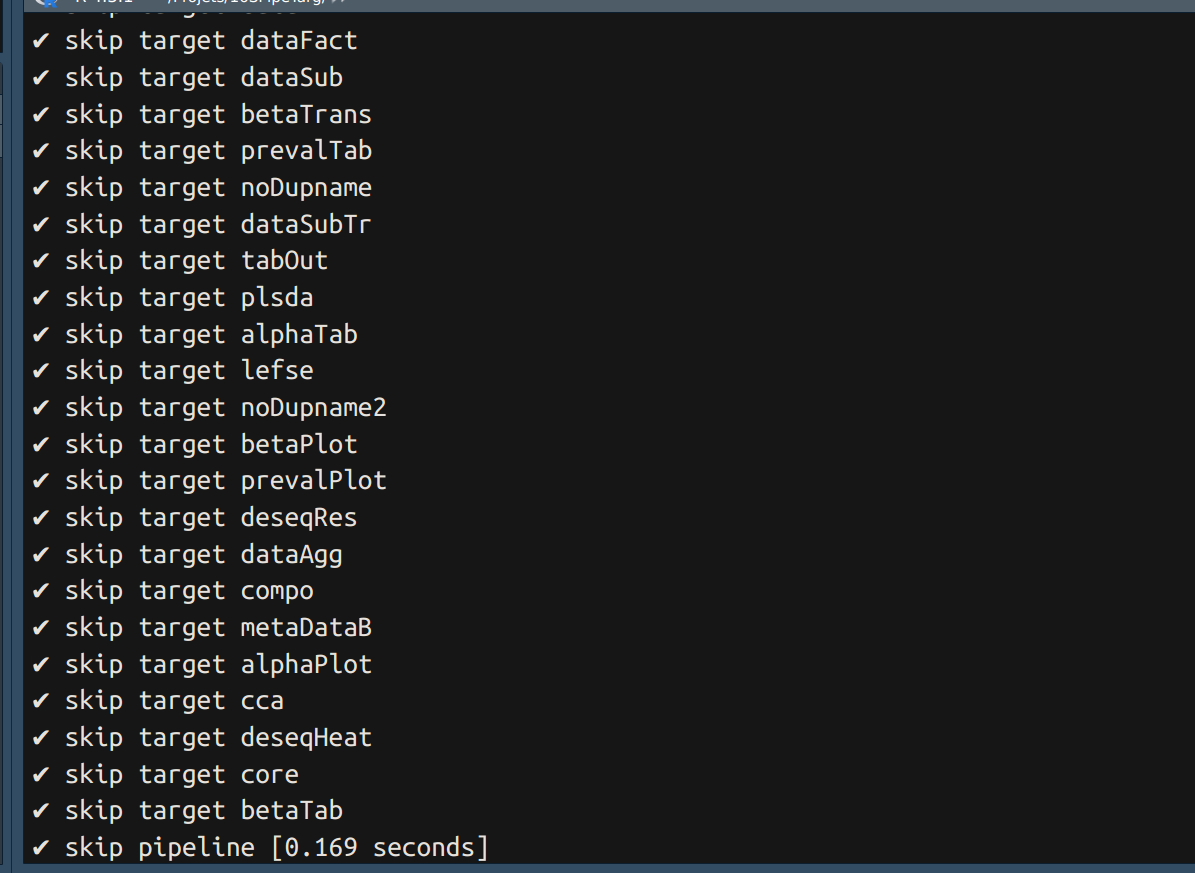
Visualisation du pipeline
La fonction tar_visnetwork() affiche un DAG du pipeline au temps t, mettant en évidence l’état des étapes (done, waiting, error).

Visualisation des objets de sortie
Contrairement à une utilisation classique de R, les objets ne sont pas stockés dans l’environnent global mais dans les dossiers _targets > objects. Il s’agit de fichiers compilés lisibles uniquement par targets via la commande tar_read(object):
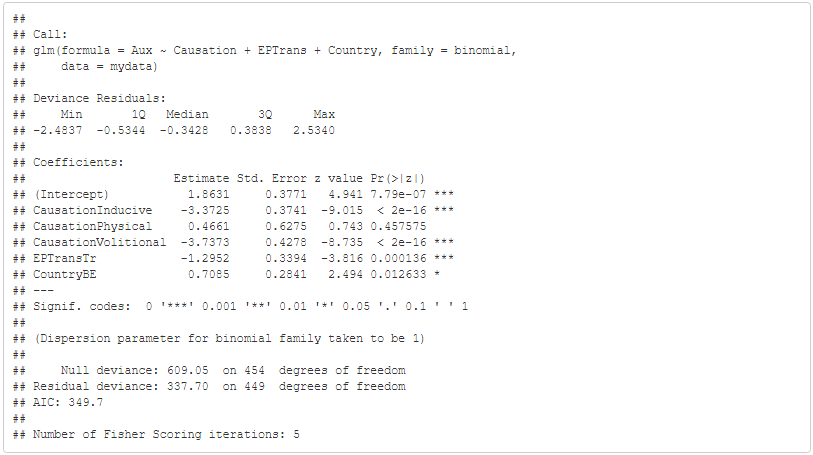
Résultats
Appeler les objets de sortie de targets dans les chunks d’un fichier qmd:
ou
Intégrer quarto au pipeline
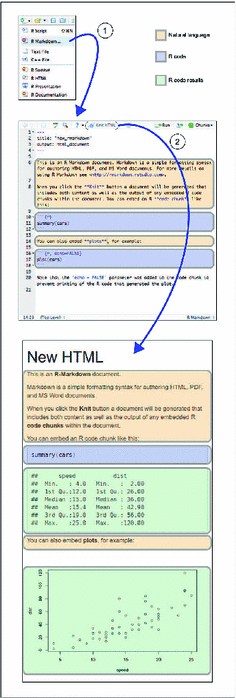
Il est possible de générer un document quarto avec la fonction quarto_render du package quarto.
- générer un rapport lors d’une étape du pipeline avec :
Controler ses supports de communication

RMarkdown
Unifier en unique document contexte, code, résultat, interprétation pour assure la cohérence des analyses …
quarto
- Successeur de
Rmarkdown - Multi langages (R, Python, Julia, Observable)
- Documents de type rapports paginés, documents HTML, sites web, livres, slides
- Interactivité
- Export en
html,pdf,docx,ePub, …
An open-source scientific and technical publishing system
Quarto - exemples
---
title: "Quarto Computations"
---
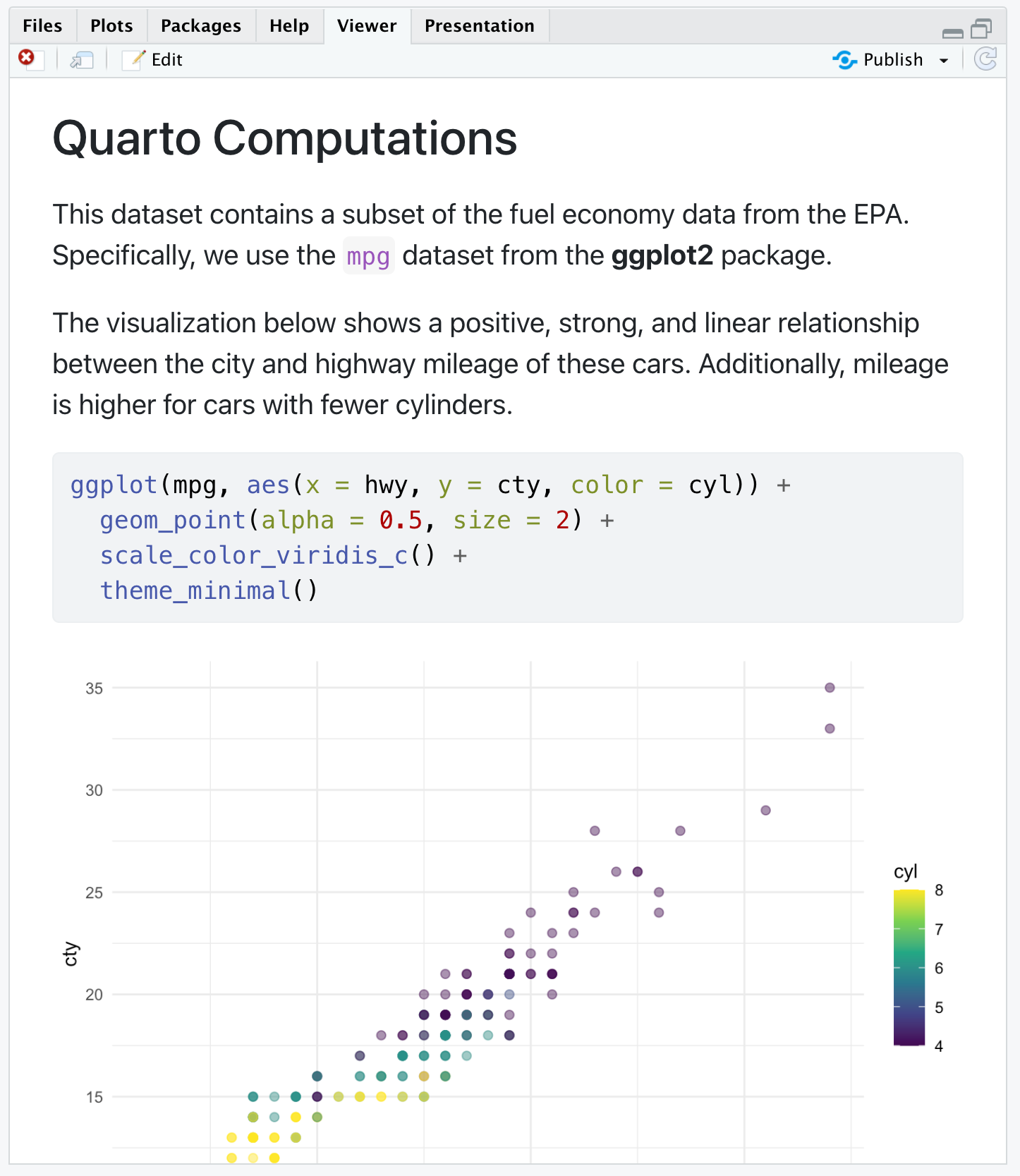
This dataset contains a subset of the fuel economy data from the EPA.
Specifically, we use the `mpg` dataset from the **ggplot2** package.
```{r}
#| label: load-packages
#| echo: false
library(ggplot2)
```
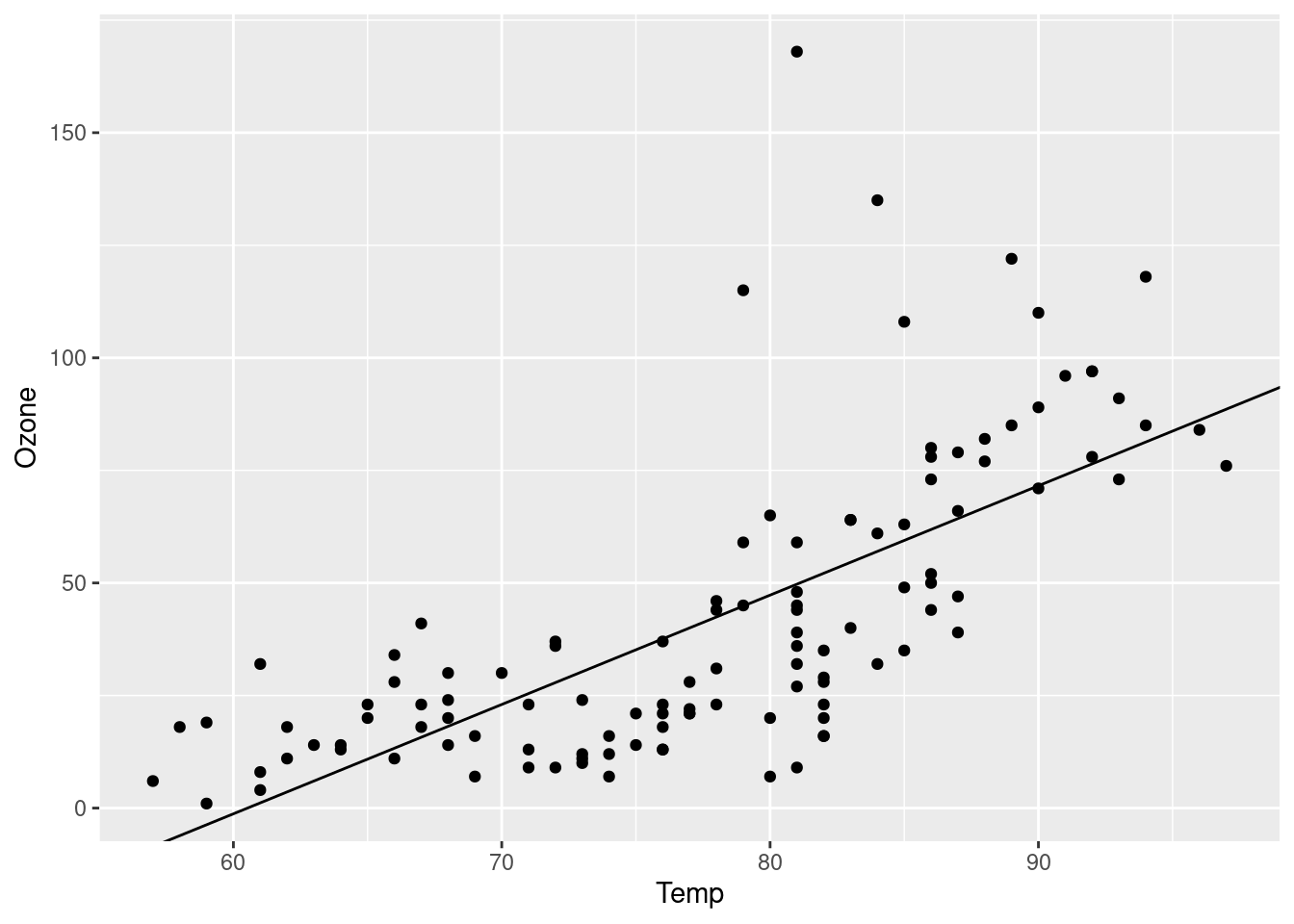
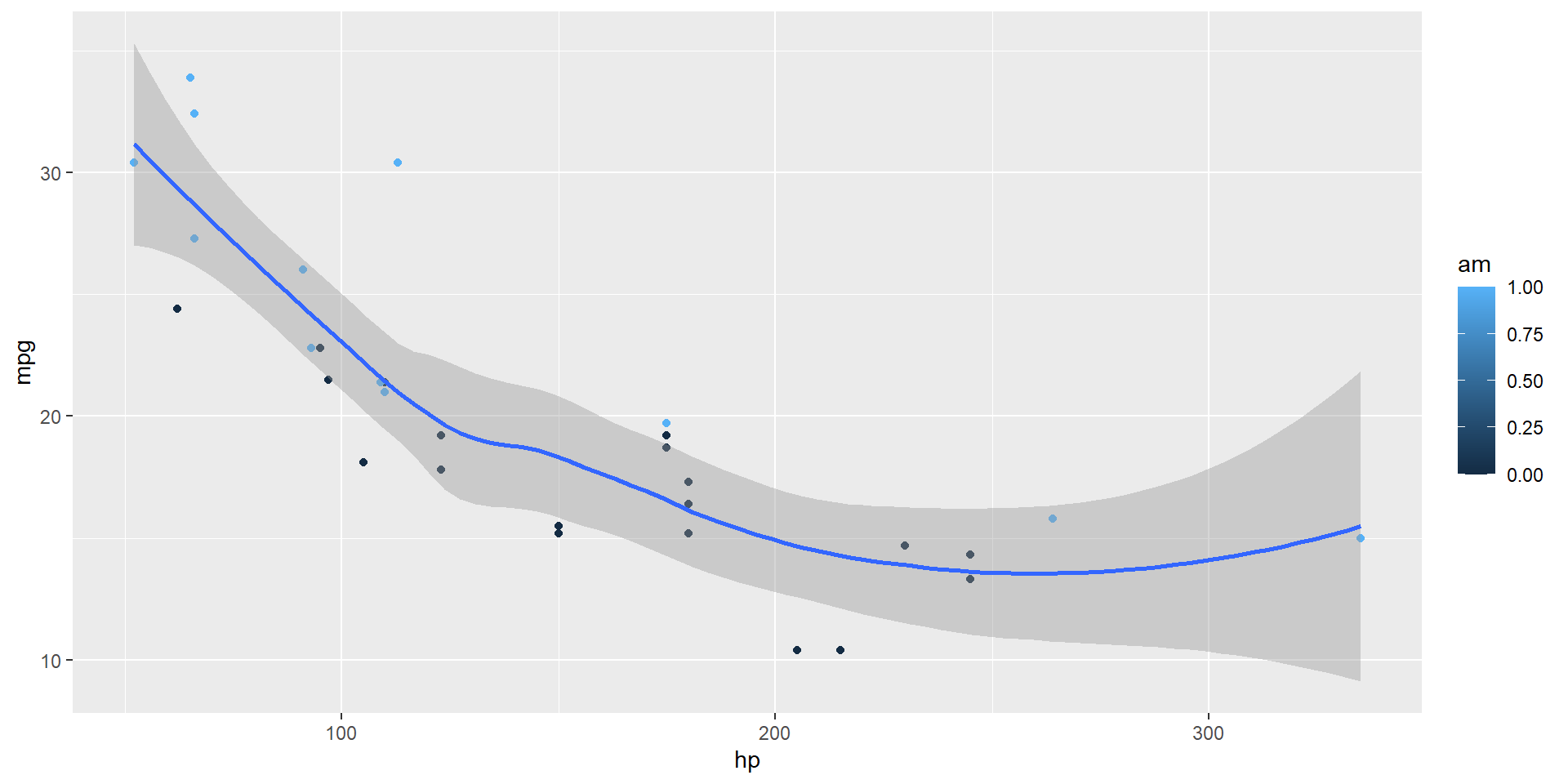
The visualization below shows a positive, strong, and linear relationship between the city and highway mileage of these cars.
Additionally, mileage is higher for cars with fewer cylinders.
```{r}
#| label: scatterplot
ggplot(mpg, aes(x = hwy, y = cty, color = cyl)) +
geom_point(alpha = 0.5, size = 2) +
scale_color_viridis_c() +
theme_minimal()
```
Quarto facilite le passage d’un format à l’autre
Les nouveautés de quarto (HTML)
- YAML standardisé entre les formats
- Decouplé de R et RStudio
- Présentation plus cohérente entre les formats
- Tab Panels
- Code Highlighting
- Mise en cache des sorties (freezing)
- Mise en page précise
RMarkdown
Quarto
Les options sont déplacées au sein du chunk avec #| (hash-pipe) pour chaque ligne. Transition facilitée avec knitr::convert_chunk_header()
Code highlighting
Widgets
Interactivité
Diffusion en CI/CD : GitLab Pages
.gitlab-ci.yml
# The Docker image that will be used to build your app
image: rocker/verse:4.2
# Functions that should be executed before the build script is run
before_script:
- quarto install extension davidcarayon/quarto-inrae-extension --no-prompt
pages:
script:
- quarto render
artifacts:
paths:
# The folder that contains the files to be exposed at the Page URL
- public
rules:
# This ensures that only pushes to the default branch will trigger
# a pages deploy
- if: $CI_COMMIT_REF_NAME == $CI_DEFAULT_BRANCHGithub Actions for Quarto
- quarto-dev/quarto-actions/setup - Install Quarto
- quarto-dev/quarto-actions/render - Render project
- quarto-dev/quarto-actions/publish - Publish project
Et si on mélange tout ça
- Mettre en place un PGD, une gestion des métadonnées et un vocabulaire contrôlé
- Déposer les données sur un entrepôt de référence
- Versionner le code avec
gitet le partager sur Git[Lab|Hub] - Fixer l’OS et les dépendances avec
dockeret les packages avecrenv - Contrôler le workflow d’analyse avec
targets - Rédiger les documents avec
quarto - Déporter les calculs grâce à l’intégration continue
- Déployer les documents avec GitLab Pages
Step by step

ToDo
- Mettre en place un PGD, une gestion des métadonnées et un vocabulaire contrôlé
- Déposer les données sur un entrepôt de référence
- Versionner le code avec
gitet le partager sur Git[Lab|Hub] - Fixer l’OS et les dépendances avec
dockeret les packages avecrenv - Contrôler le workflow d’analyse avec
targets - Rédiger les documents avec
quarto - Déporter les calculs grâce à l’intégration continue
- Déployer les documents avec GitLab Pages
Step-by-step
- Essayer de nouvelles choses progressivement
- Tester sur un petit projet pour commencer
- Se documenter et rester au courant des nouveautés. Les techno évoluent et facilitent l’usage
- Échanger avec les collègues, la communauté, le PEPI
- Rester pragmatique
- Adapter l’usage aux besoins
Biblio
1. Quintana D. Five things about open and reproducible science that every early career researcher should know. Open Science Framework. 2022. doi:10.17605/OSF.IO/DZTVQ.
2. The Turing Way Community. The turing way: A handbook for reproducible, ethical and collaborative research. 2022. doi:10.5281/ZENODO.3233853.
3. Peng RD. Reproducible research in computational science. Science. 2011;334:1226–7.
4. Wickham H, Çetinkaya-Rundel M, Grolemund G. R for data science. O’Reilly Media; 2023. https://r4ds.hadley.nz/.
5. CIRAD-DGDRS-DIST-FRA, editor. Le cycle de vie des données. Intégrer la gestion de données scientifiques aux activités de recherche. 2017. https://agritrop.cirad.fr/594579/.
6. Sébire F. Check-list de l’Institut Pasteur pour des bonnes pratiques de gestion des données de recherche. 2023. https://hal.science/hal-04123336.
7. Bhandari Neupane J, Neupane RP, Luo Y, Yoshida WY, Sun R, Williams PG. Characterization of leptazolines a–d, polar oxazolines from the cyanobacterium leptolyngbya sp., reveals a glitch with the “willoughby–hoye” scripts for calculating NMR chemical shifts. Organic letters. 2019;21:8449–53.
8. Russo F, Righelli D, Angelini C. Advantages and limits in the adoption of reproducible research and r-tools for the analysis of omic data. Cham: Springer International Publishing; 2016.

PEPI IBIS – 15 septembre 2023

Comment ça marche ?