Élément du portfolio - projet METABARFOOD
Visite HCERES
16 décembre 2024
Le métabarcoding
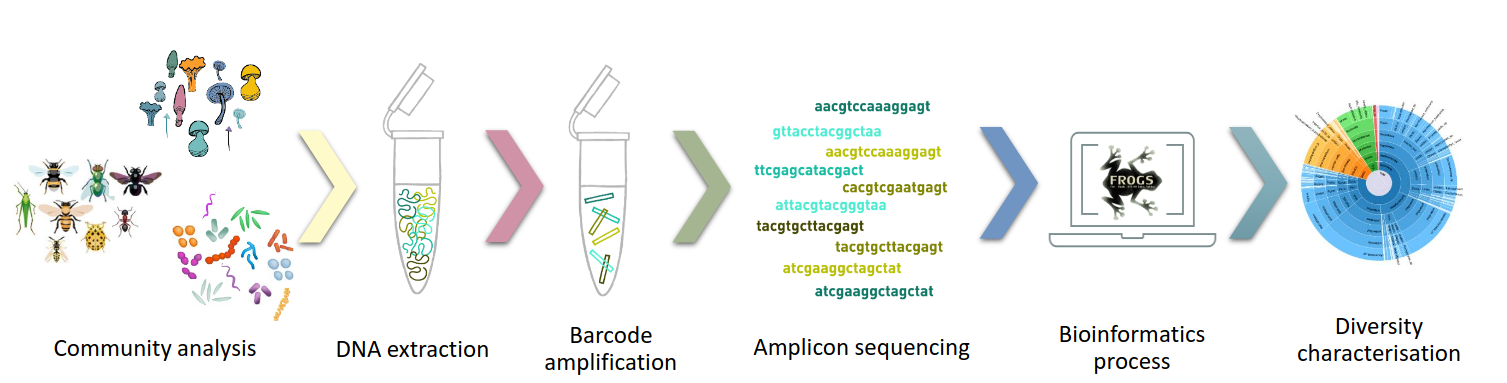
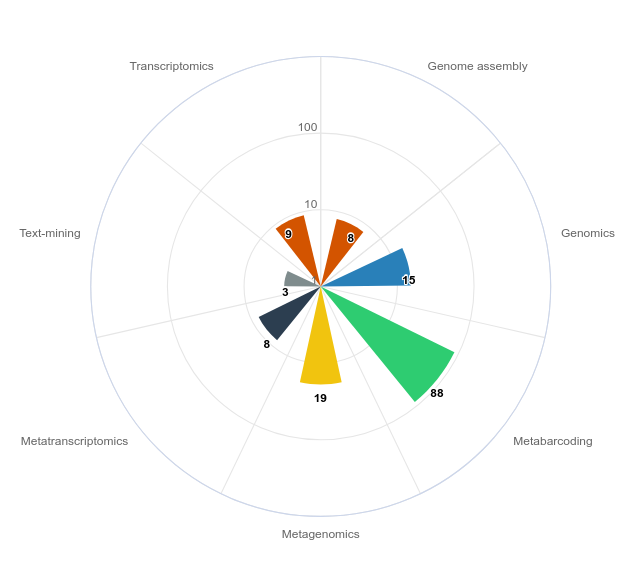
- Migale possède une expertise reconnue en analyse de données de métabarcoding
Le projet METABARFOOD
- Projet financé Métaprogramme MEM (Méta-Omiques des Écosystèmes microbiens - 2015) porté par Delphine Sicard (SPO, INRAE Montpellier)
-
Partenaires INRAE
- LUBEM (Université Brest), SPO (Montpellier), SayFood (Grignon/Saclay), Migale (Jouy-en-Josas)
-
Objectifs
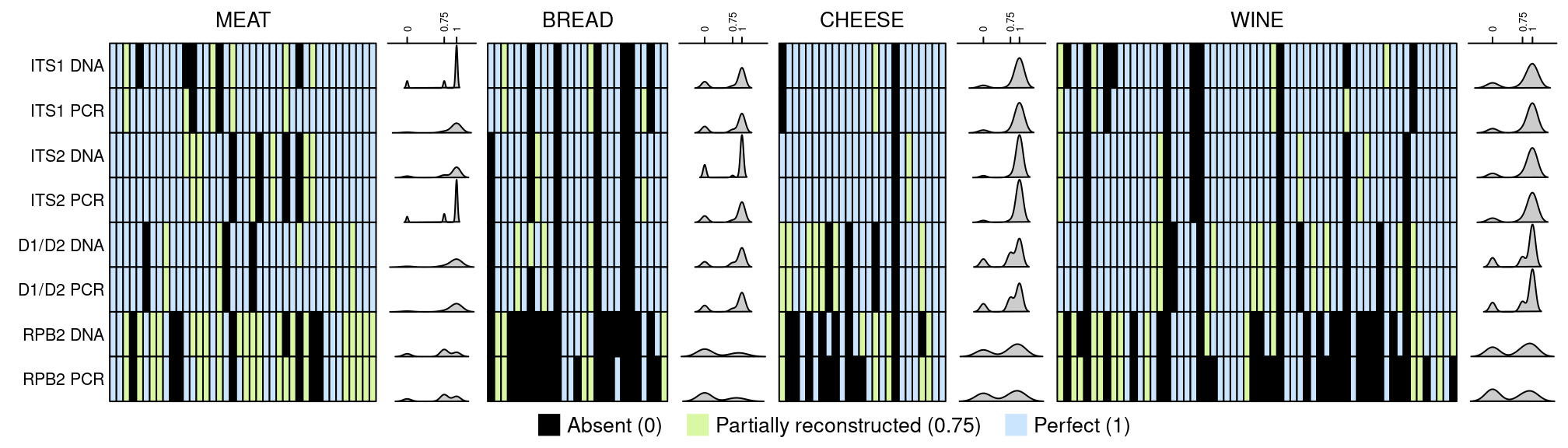
- Comparer les pipelines de métabarcoding et les marqueurs (ITS1, ITS2, D1/D2, RPB2) pour déterminer la diversité fongique dans différentes matrices alimentaires (Pain, Viande, Vin, Fromage)
- Tester ces approches sur des jeux de données synthétiques et réels
-
Échantillons
- 96 échantillons MOCKs (27-60 espèces) : 4 matrices x 4 marqueurs x 2 conditions (PCR/DNA) x 3 réplicats
- 144 Échantillons réels : 4 matrices x 4 marqueurs x 3 échantillons x 3 réplicats
- Séquençage Illumina Miseq 2x250 (GeT-PlaGe, INRAE Toulouse)
-
Contexte
- Financement de la génération des données
- Délai entre le début du projet et l’acquisition des données
- Projet identifié comme une opportunité intéressante pour Migale
De nombreux résultats issus du projet
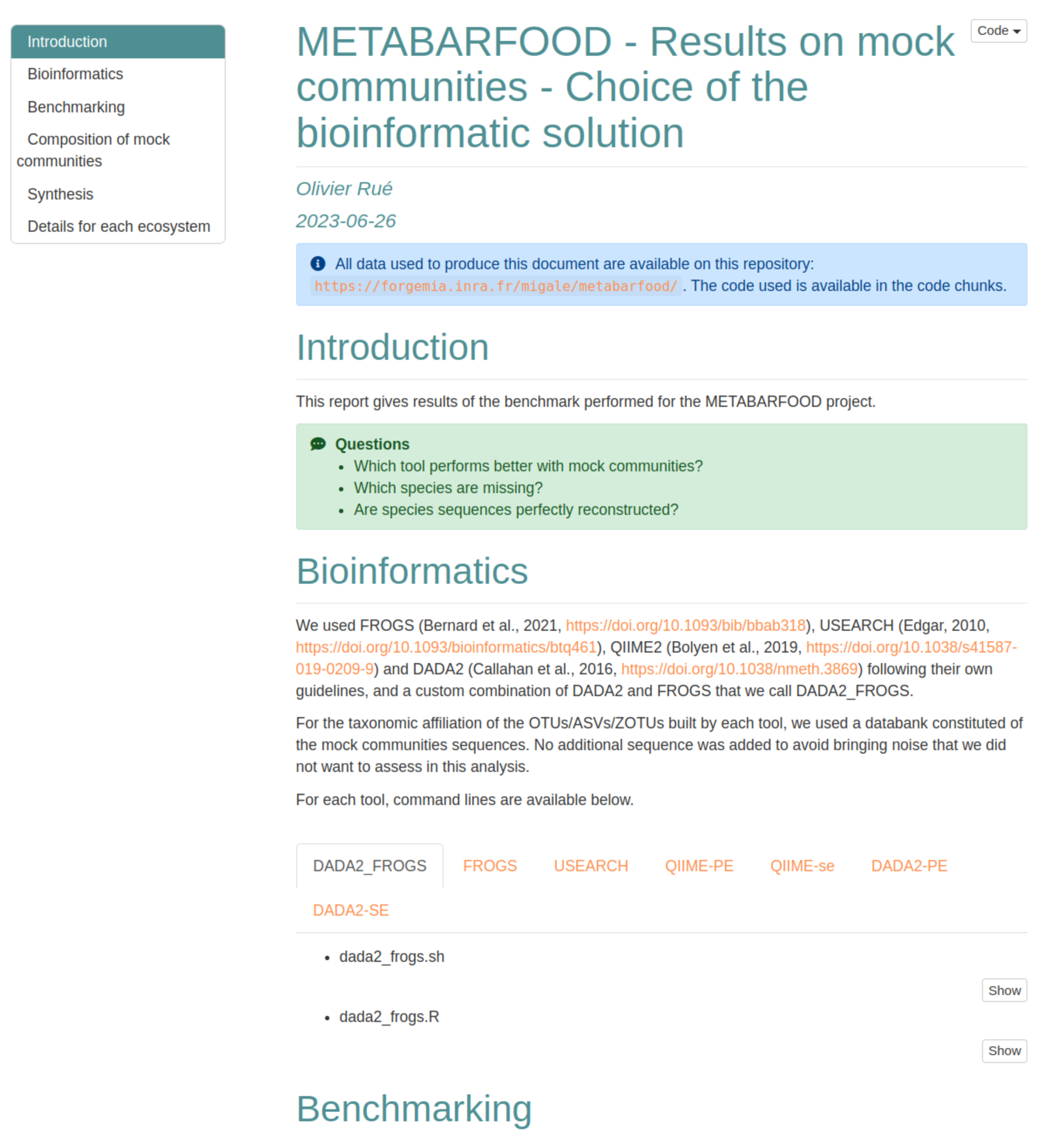
- Bioinformatique
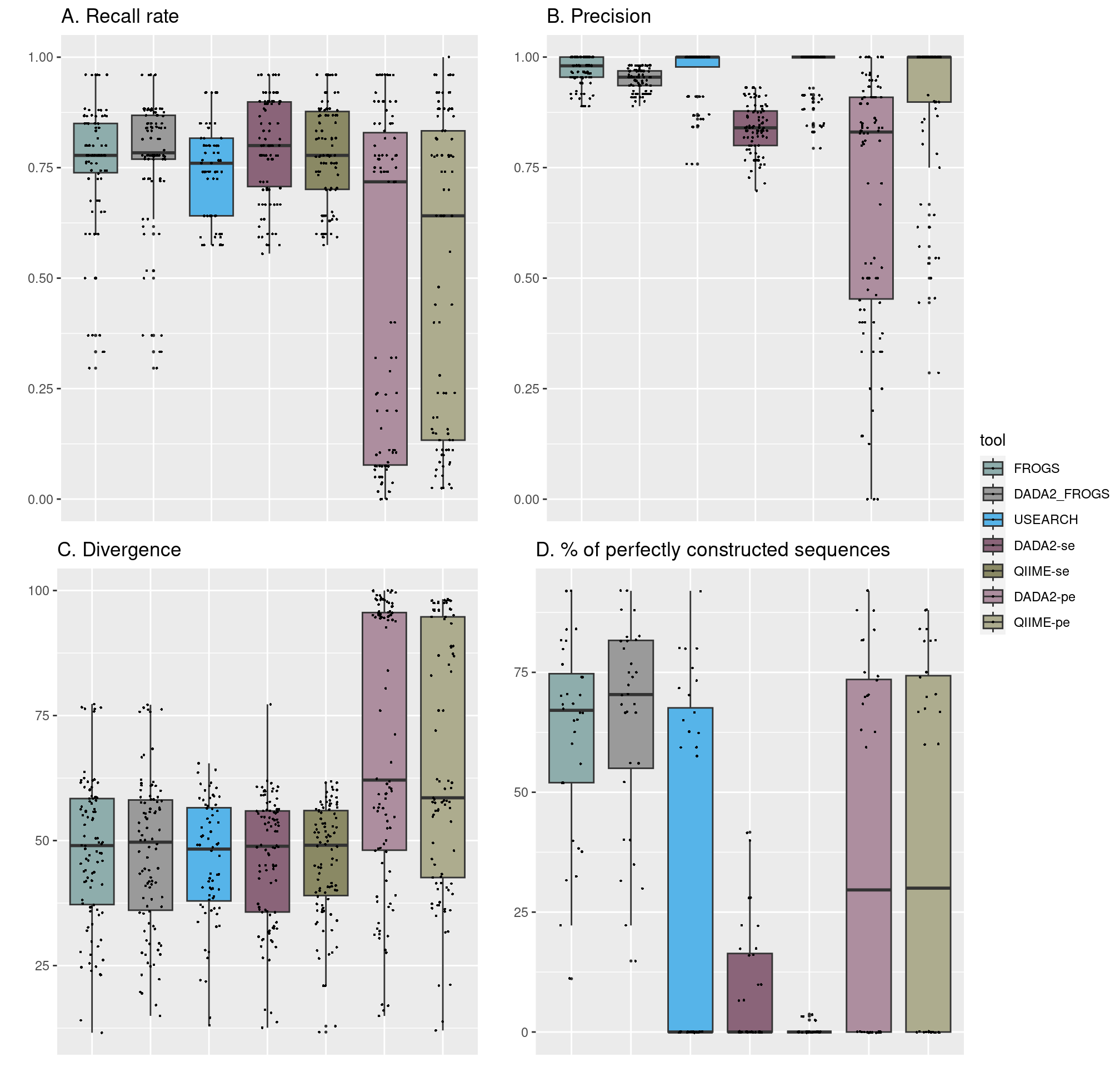
- Benchmark des outils bioinformatiques
- Données utilisées dans la publication de FROGS-ITS [1]
- Développement d’une approche mixte DADA2+FROGS plus performante
- Scientifique
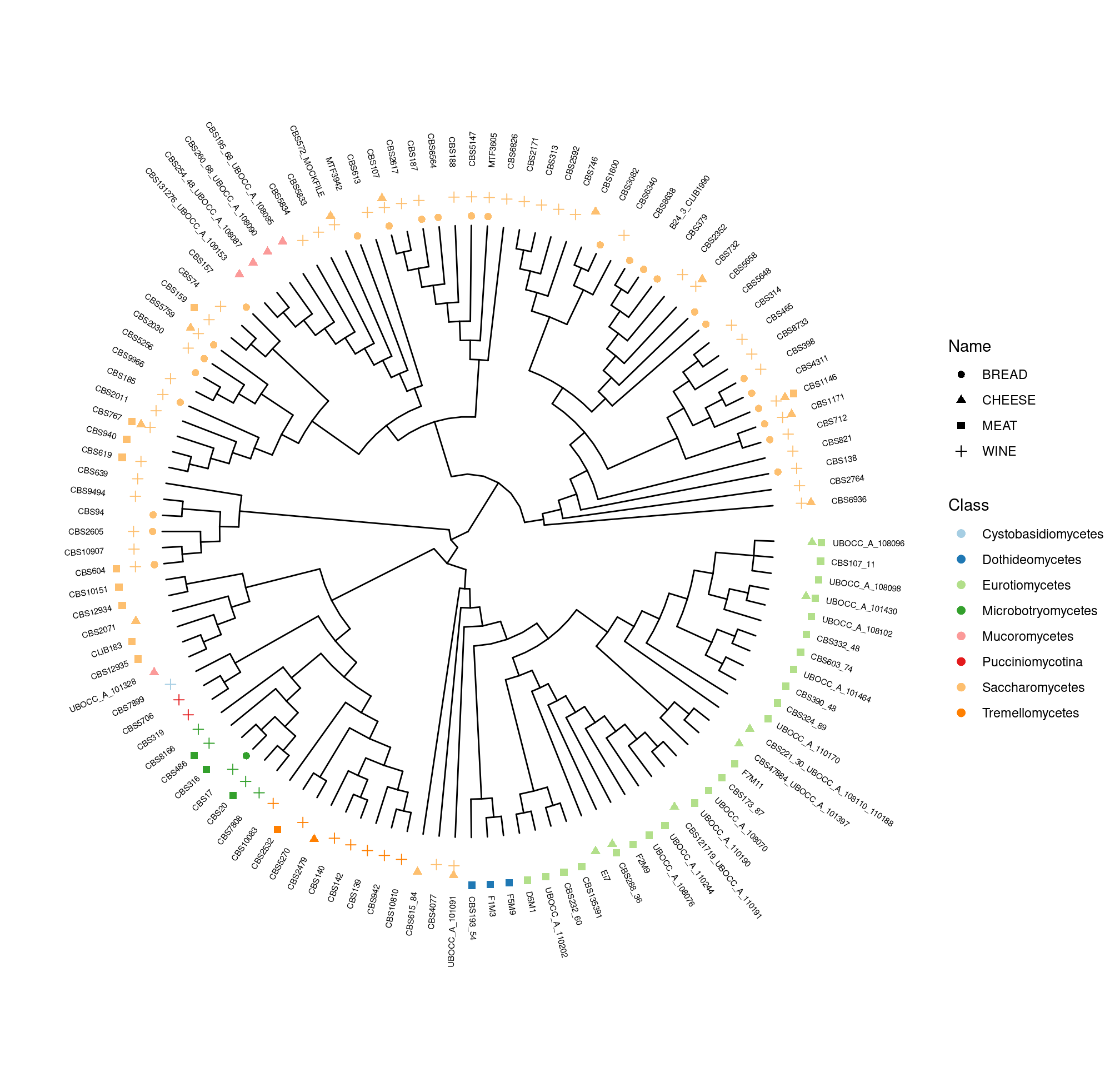
- Création d’une banque de référence des 4 marqueurs pour les souches d’intérêt (472 au total)
- Évaluation des marqueurs sur données synthétiques et réelles
- Méthodologique
- Analyse des échantillons réels a nécessité la curation manuelle de 2,359 ASVs
- Processus itératif mis en place sur tous les projets

Les principes FAIR appliqués au projet
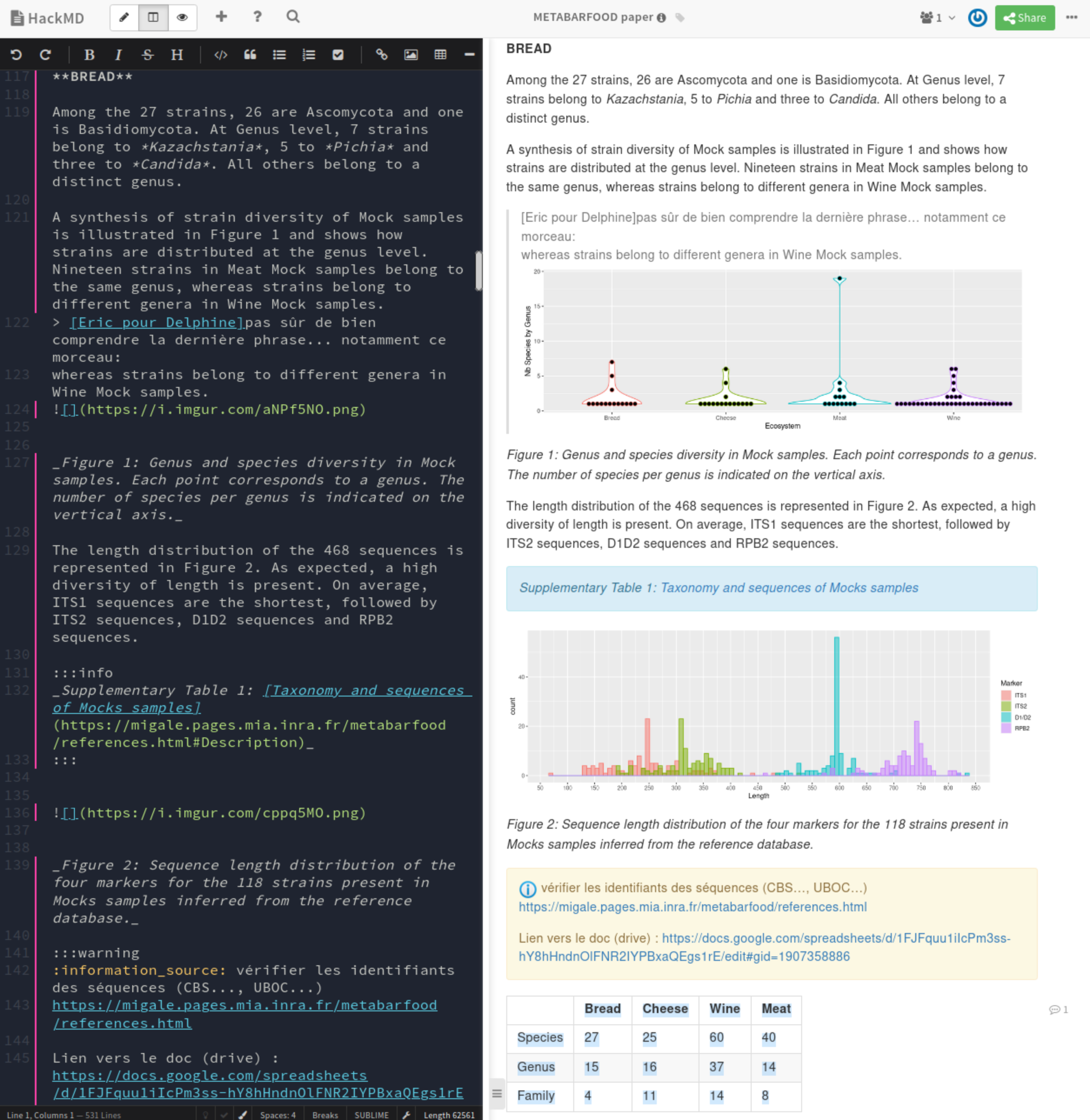
- Rapports d’analyse en ligne, complets et évolutifs
- Dépôt du code sur forgeMIA
- déploiement continu des rapports d’analyse
- Dépôt du code sur forgeMIA
- Analyses reproductibles, travail facilité lors de la review (banque à mettre à jour)
- Mise en place d’outils collaboratifs
- pour faciliter les échanges à distance et la rédaction de l’article
- Mise à disposition du code
- Code archivé sur HAL ↔︎ Software Heritage
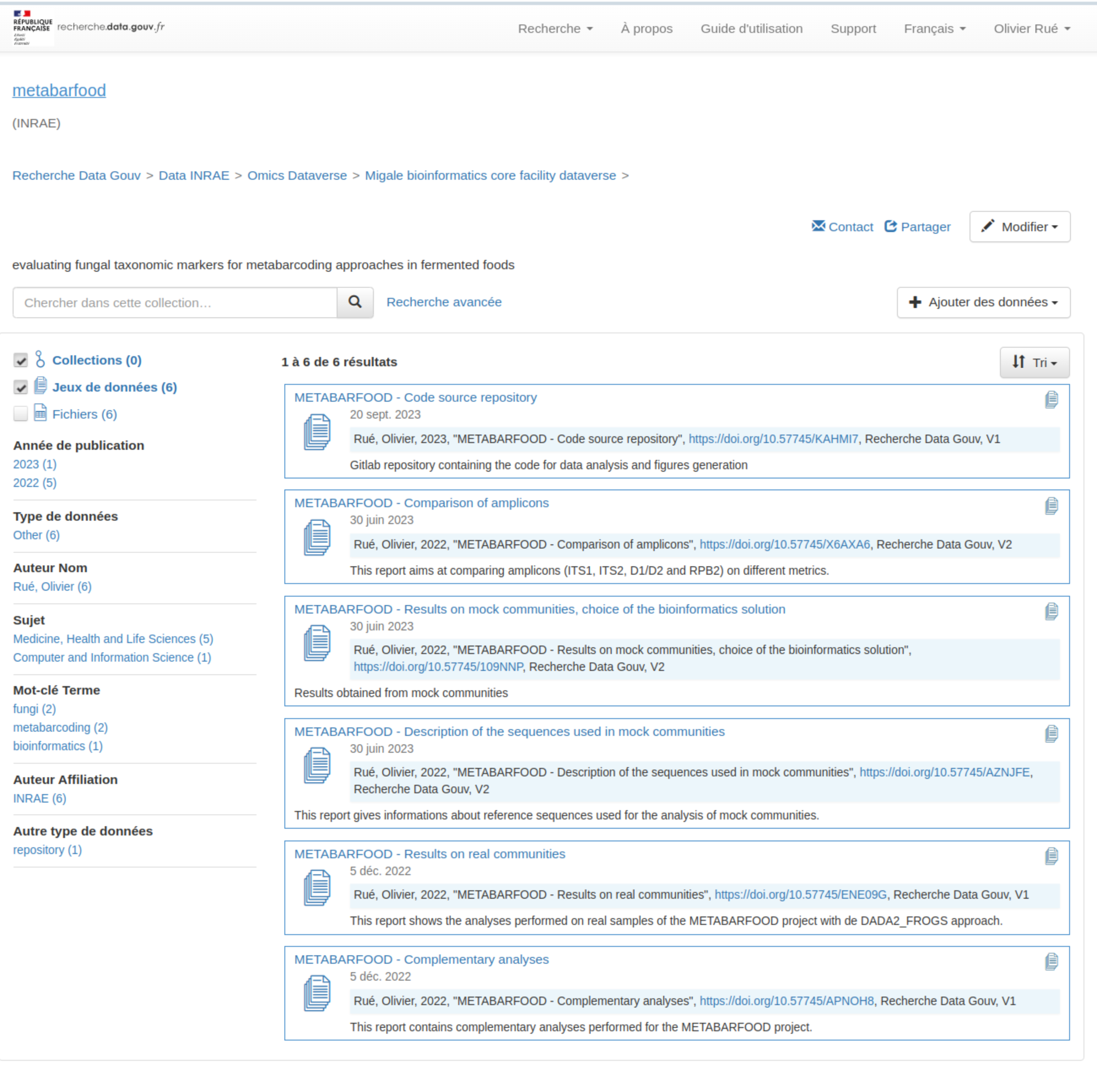
- Mise à disposition des données (brutes et intermédiaires)
- Dataverse dédié
- Valorisation des résultats
- Travail publié dans Peer Community Journal en 2023 [2]

Conclusion
- Projet…
- mené à son terme,
- valorisé
- et sur lequel on a capitalisé
-
Nouvelles ressources
à la fois bioinformatique …
- Méthodologie intégrée dans FROGS v5.0.0 et aux formations associées
- et scientifique …
- Résultats disponibles pour la communauté
- … en appliquant les principes FAIR, en se basant sur les ressources institutionnelles
Merci de votre attention
References
1. Bernard M, Rué O, Mariadassou M, Pascal G. FROGS: a powerful tool to analyse the diversity of fungi with special management of internal transcribed spacers. Briefings in Bioinformatics. 2021;22. doi:10.1093/bib/bbab318.
2. Rué O, Coton M, Dugat-Bony E, Howell K, Irlinger F, Legras J-L, et al. Comparison of metabarcoding taxonomic markers to describe fungal communities in fermented foods. Peer Community Journal. 2023;3.

Visite HCERES