Plateforme Migale
Visite départements MICA/MathNum
27 septembre 2023
Rappel des missions de la plateforme
- Mettre à disposition une infrastructure de calcul scientifique pour la génomique
- Calcul / Stockage / Outils / Données
- Diffuser un savoir-faire en bioinformatique et biostatistique
- Formations / Assistance / Conseil
- Concevoir et développer des applications
- Développement et mise à disposition d’outils / Interfaces innovantes
- Accompagnement dans l’analyse de données
- Génomique / Génomique comparée / Métagénomique / Text Mining
Équipe

- 6,35 ETP permanents répartis sur 10 personnes
- Départ Damien Berry en février 2022
- Arrivée de Christelle Hennequet-Antier en février 2022
- Passage de Sophie Schbath à 50% en juin 2022
- 2 postes non pourvus sur la période 21-23
- IE Bioinformaticien (MICA, Mobilité, 2021)
- IE Admin Système (MICA, concours externe 2022, mobilité au fil de l’eau 2023)
Des services qui s’enrichissent
- Mise en place du service d’analyse de données (2016)
- Focus thématique : génomique et métagénomique microbienne
- Ecosystèmes variés : environnement, aliment, microbiote intestinal animal…
- Accompagnement global des utilisateurs : Outils, Expertise / Analyse, Formation
- Accent mis sur l’autonomisation par la transparence et la reproductibilité : Outils conviviaux, tutoriels, rapports réutilisables et détaillés (comment / pourquoi)
- Application du même schéma au text-mining pour la bioinformatique (depuis fin 2019)
Résultats / projets complémentaires

- 130 projets traités depuis 2016, \(\approx\) 20/an dont :
- metaPDO-Cheese (Irlinger et al, in prep)
- METABARFOOD (Rué et al., PCI Microbiology 2023)
15 modules de formations annuels, dont :
- Analyse de données métagénomiques « shotgun » (🆕 2020)
- Graphiques sous R avec ggplot2 (🆕 2020)
- Comparaison de génomes microbiens (🆕 2020)
- Développement d’une application avec Shiny (🆕 2020)
- Bonnes pratiques pour une meilleure reproductibilité de ses analyses (🆕 2021)
- Introduction au text-mining avec AlvisNLP (🆕 2022)
- Manipulation de données avec R : Introduction à Tidyverse (🆕 2023)
Le projet METABARFOOD (2015-2023)
- Projet financé Métaprogramme MEM (2015) porté par Delphine Sicard (SPO)
-
Partenaires
- LUBEM (INRAE / Université Brest), SPO (INRAE Montpellier), SayFood (INRAE Saclay), Migale (INRAE Jouy-en-Josas)
-
Objectifs
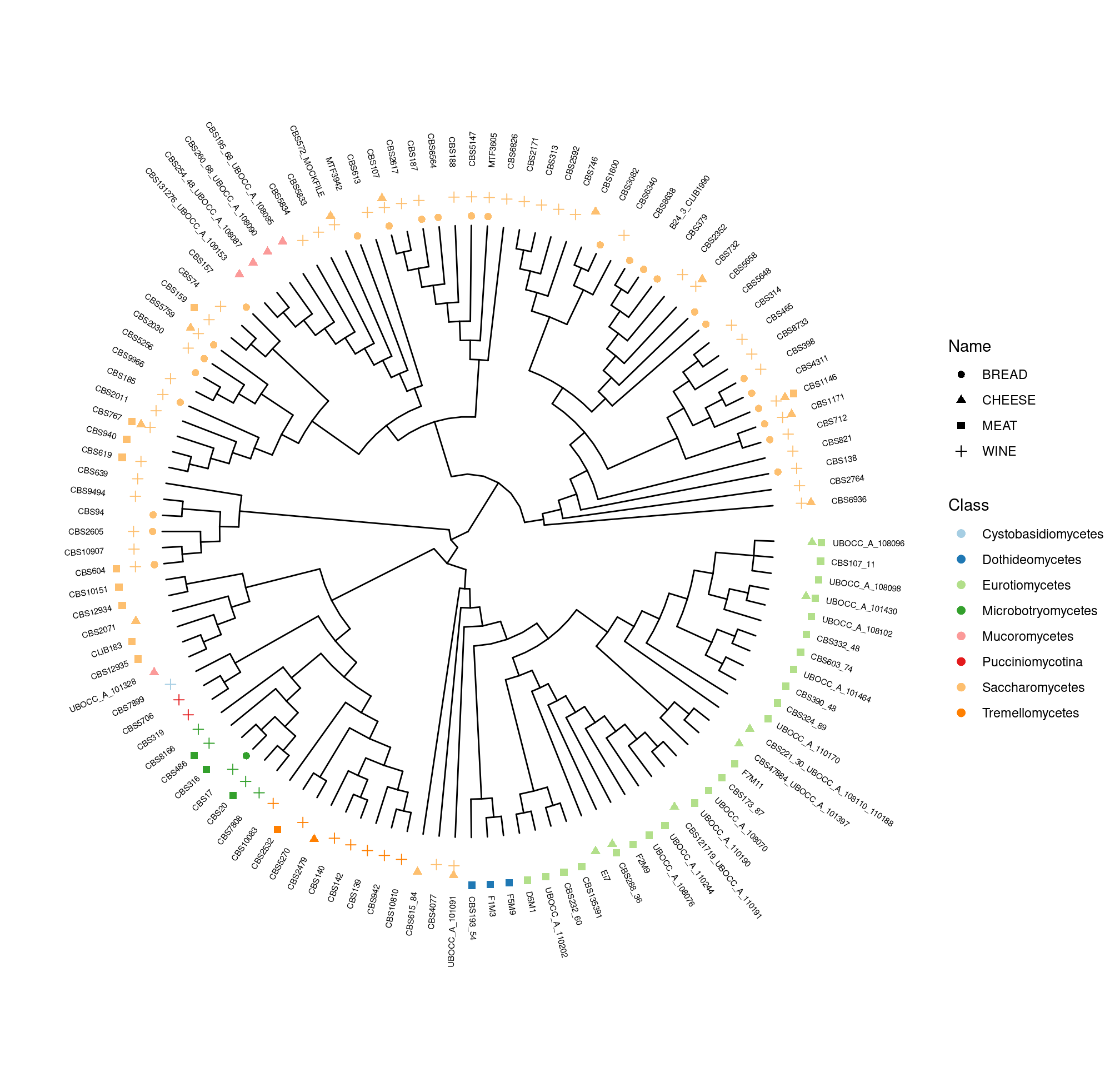
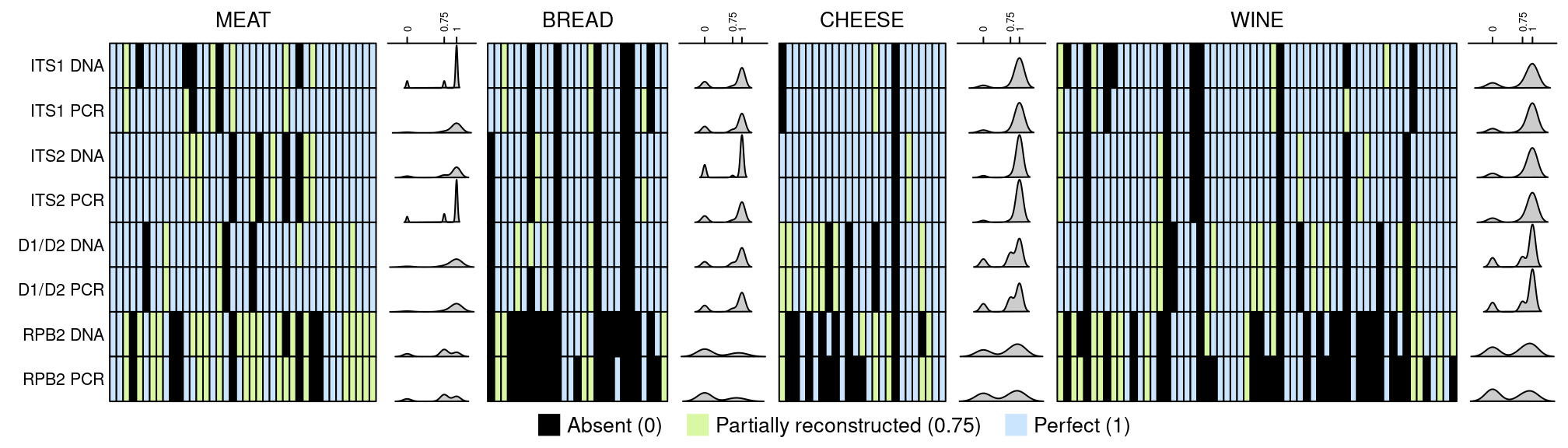
- Comparer les pipelines de métabarcoding et les marqueurs (ITS, D1/D2, RPB2) pour déterminer la diversité fongique dans différentes matrices alimentaires (Pain, Viande, Vin, Fromage)
- Tester ces approches sur des jeux de données synthétiques et réels
-
Échantillons
- 96 échantillons MOCKs (27-60 espèces) : 4 matrices x 4 marqueurs x 2 conditions (PCR/DNA) x 3 réplicats
- 144 Échantillons réels : 4 matrices x 4 marqueurs x 3 échantillons x 3 réplicats
- Séquençage Illumina Miseq 2x250 (GeT-PlaGe Toulouse)
Résultats
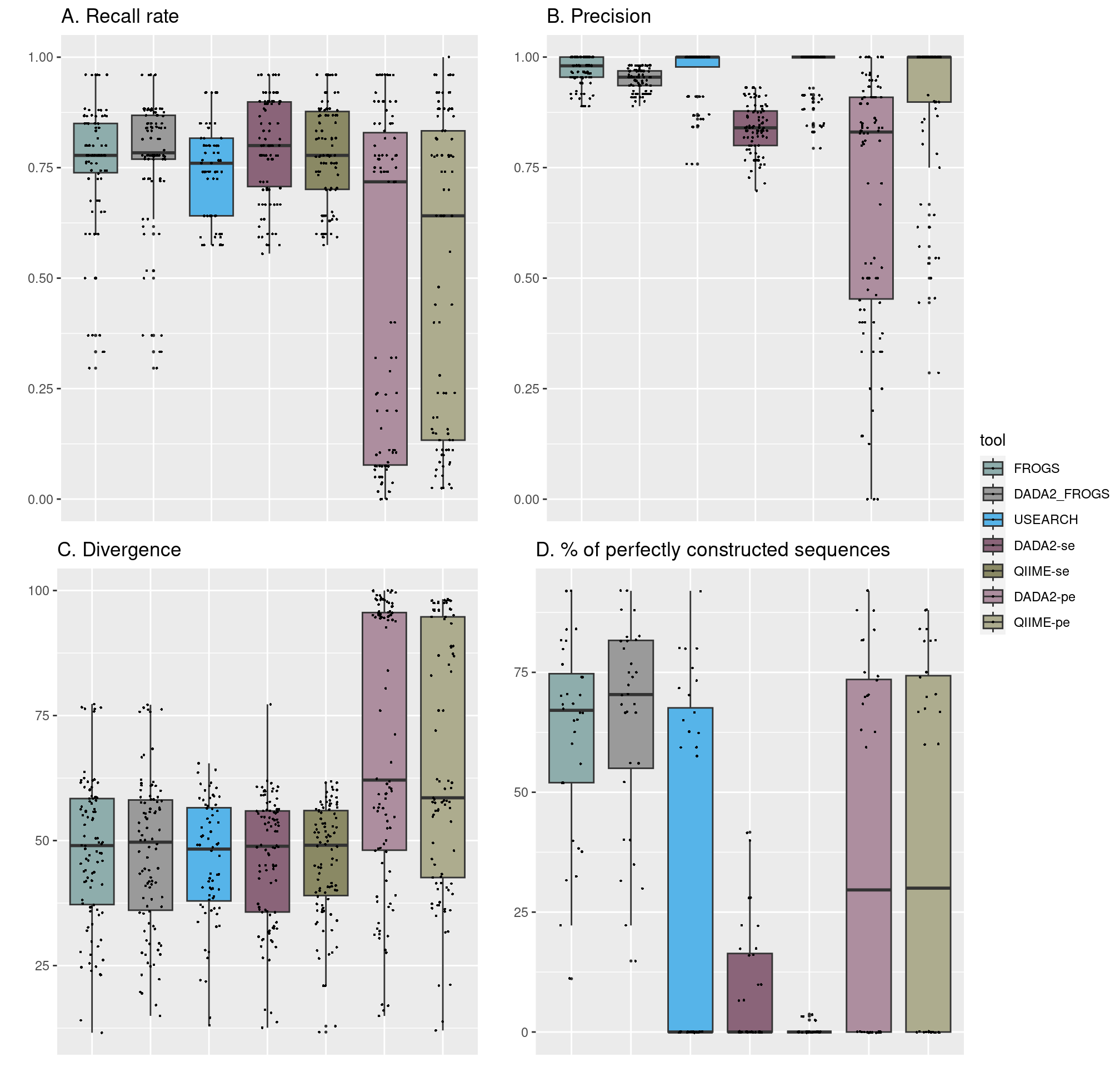
- Benchmark des outils bioinformatiques
- Développement d’une approche mixte DADA2+FROGS plus performante
- Création d’une banque de références de 472 références de souches d’intérêt
- Évaluation des marqueurs sur données synthétiques et réelles
- Analyse des échantillons réels
- curation manuelle de 2,359 ASVs
- généralisation des conclusions sur les marqueurs
Un nouvel élan après le COVID
- Reprise du leadership pour mobiliser les troupes
- Mise en place d’outils collaboratifs
- Dépôt du code sur forgeMIA
- déploiement continu des rapports d’analyse
- analyses reproductibles, travail facilité lors de la review (banque à mettre à jour)
- Dataverse dédié / Code archivé sur HAL ↔︎ Software Heritage
- DADA2 en cours d’intégration dans FROGS et ses formations
- Travail publié dans Peer Community Journal en 2023

Conclusion
- Projet mené à son terme et valorisé
- Nouvelles ressources à la fois bioinformatique et scientifique
- Mise en place des principes FAIR en se basant sur les ressources institutionnelles
- Outils déployés par la plateforme adaptés aux projets collaboratifs
Positionnement : De la caractérisation de la biodiversité des aliments…
Projets
- METABARFOOD (MICA, MEM 2015)
- METAPDO Cheese (MICA, France Génomique 2017)
- REDLOSSES (MICA, ANR 2017)
- 36 projets “FOOD” menés dans le cadre du service d’analyse
Développements et Résultats
- Ajout de fonctionnalités dans FROGS (ITS, DADA2,… )
- Omnicrobe : base de données d’habitats et phénotypes microbiens (en collab. avec Bibliome et StatInfOmics)
- Évolution de la formation metabarcoding
- 15 publications en collaboration
…à la caractérisation fonctionelle des aliments fermentés et l’écologie sythétique
Projets
- metaSimFood (MICA, ANR 2022)
- DOMINO (MICA, Horizon Europe 2023)
- MuDiS4LS (IFB, PIA3 Equipex+ (Co-coordination IS5 Holobionte))
- FdF (INRAE, France2030 2022)
Développements et Résultats
- Gestion des données (dont DMP)
- Analyse de données métagénomiques shotgun :
- expertise
- procédures d’analyses
- formations
- Développement de l’entrepôt de FdF :
- matériel
- développement logiciel
- outils d’analyses standardisés
- formations
Implication dans l’ensemble du projet : infrastructure, gestion des données, analyse bioinfo et analyses statistiques.
Ex : projet SoyUses (MICA, France 2030, AAP PROT-LEG 2023, soumis)
Merci de votre attention

Visite départements MICA/MathNum