Introduction aux bonnes pratiques pour des analyses reproductibles
Module 25
March 21, 2024
Introduction
Informations pratiques
- 9h00 - 17h00
- 2 pauses
- Déjeuner au restaurant INRAE (si vous le souhaitez)
- Questions bienvenues
- Chacun a quelque chose à apprendre des autres
Diffusion des documents
Tous les documents présentés lors de cette formation sont destinés à être distribués et sont disponibles sur https://documents.migale.inrae.fr
Tour de table
- Qui êtes vous ?
- Institution, laboratoire, métier …
- Quelles sont vos approches actuelles concernant la reproductibilité de vos analyses ?
- Connaissez vous le sigle FAIR ?
- Avez vous déjà été confrontés à des soucis de reproductibilité d’analyses (les votres, celles d’autres personnes …) ?
- Comment vous sentez vous ? OK / KO
Programme
- Intro : Quelques généralités sur la reproductibilité et la démarche science ouverte
- Organiser son espace de travail
- Versionner ses documents
- TP
- Utilisation de documents computationels – Notebook
- TP
- Aller vers le FAIR et rédiger son PGD
- Conclusion, discussion et aller + loin
Reproductibilité et Science ouverte
Tout le monde a déjà eu cette expérience
Un article interessant

Un matériel et méthodes décevant

Crise de la reproductibilité
Problème général, “Reproducibility Crisis”
- Remis en avant par les science sociales, notamment la psychologie
- Étendu à l’ensemble des disciplines scientifiques
Mais un problème qui n’est pas nouveau
- Expériences de la pompe à vide au XVIIe siècle (von Guericke et Boyle)

Et en bioinfo ?
Un problème vieux comme la bioinformatique :
- En 2009, moins de la moitié des expériences de transcriptomique parues dans Nature Genetics ont pu être reproduites
- Sur 50 articles citant BWA en 2011, 31 ne citent ni version, ni paramètres. 26 ne donnent pas accès aux données sous-jacentes
- Selon un sondage mené en 2016 auprès de plus de 1500 biologistes
- 70% ont déjà éprouvé des difficultés à reproduire une analyse [1]
- Ten Years Reproducibility Challenge : êtes vous capables de refaire vos analyses d’il y a 10 ans ?
Quelles sont les difficultés ?
- Problèmes d’accès aux données :
- le fameux “data available upon request”
- données brutes disponibles, mais méta-données inexistantes ou insuffisantes
- Problèmes d’accès aux outils :
- outils anciens ou obsolètes
- difficultés à installer
- Problèmes de paramétrage de l’analyse
- version des outil
- paramètres des outils
- enchaînement des outils
- Problème d’accès aux ressources nécessaires
- calcul
- stockage
Réplication ≠ Reproductibilité
La réplication indépendante d’expériences est à la base de la méthode scientifique.
En complément de réplication indépendante ( expérimentation, échantillonnage, analyse, …), la reproduction d’analyse est indispensable à l’évaluation et à la compréhension de la démarche employée.
- Il existe une ambiguïté en anglais entre réplication (réplication) et reproduction (reproducibility). Derrière la reproducibility crisis on mélange les deux :
- Impossibilité de répliquer des résultats de façon indépendante (psychologie, médecine, biologie…)
- Impossibilité de reproduire des analyses à partir des mêmes données de départ
Chacun peut déjà, par la mise en place de pratiques simples et l’utilisation d’outils conviviaux, améliorer la reproductibilité de ses travaux
Source : [2]
En pratique, qu’est ce qu’être reproductible ?
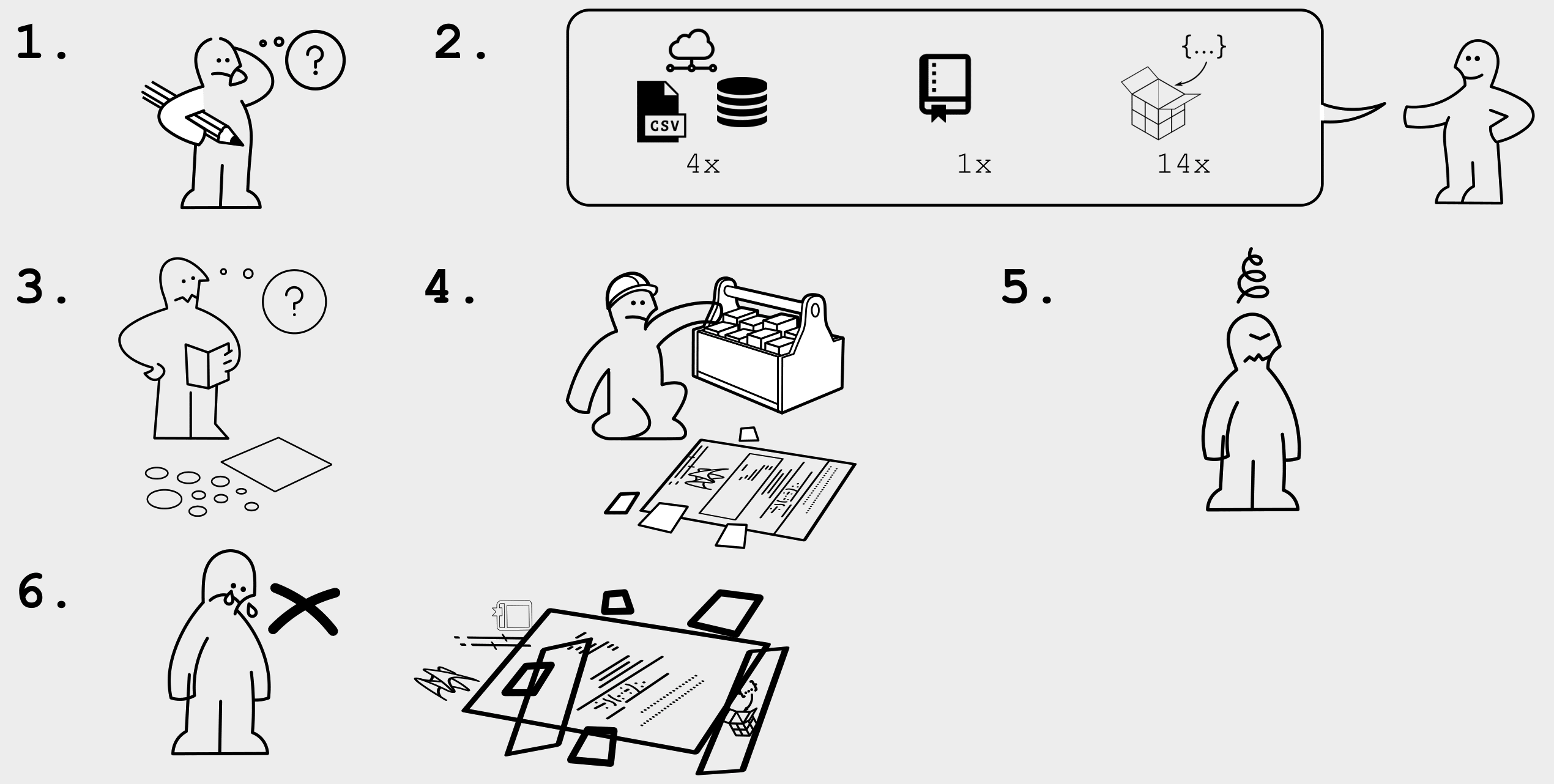
En pratique, qu’est ce qu’être reproductible (2) ?
Avoir accès :
- aux pièces (les données)
- aux outils ( les logiciels, )
- au mode d’emploi : paramètres, workflows d’analyse
Mais aussi :
- à la description des pièces, de la façon dont elles ont été produites (méta-données)
- à la documentation technique (choix techniques explicites)
- au savoir faire du monteur (formations)
- Éventuellement à un atelier équipé pour le montage (ressources informatiques)
FAIR : un pré-requis à la reproductibilité
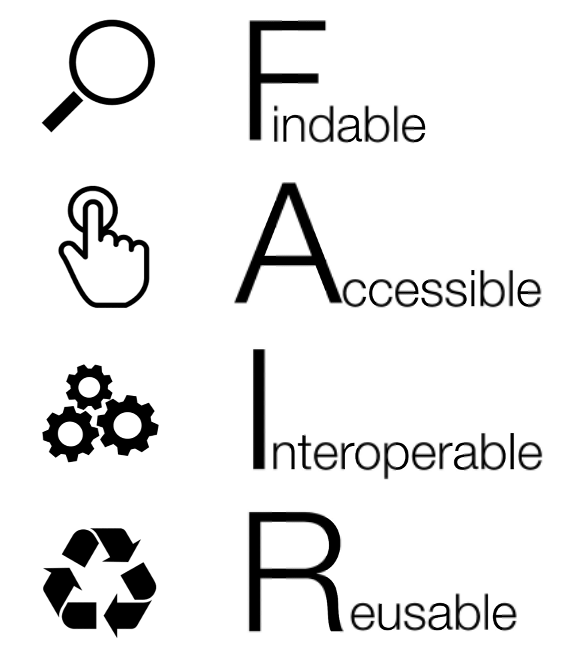
FAIR : un pré-requis à la reproductibilité
- Facile à trouver : pour les humains et les ordinateurs :
- id unique et pérennes
- métadonnées riches
- Accessibles à long terme
- entrepôt “pérenne”
- licence d’utilisation explicite ( FAIR ≠ ouvert)
- Interoperables : faciles à combiner avec d’autres jeux de données
- formats ouverts et documentés
- vocabulaire standardisé, ontologies (données et méta-données)
- Réutilisables :
- réutilisables par soi, par d’autres
- réutilisables par des machines
[3]
Le spectre de la reproductibilité
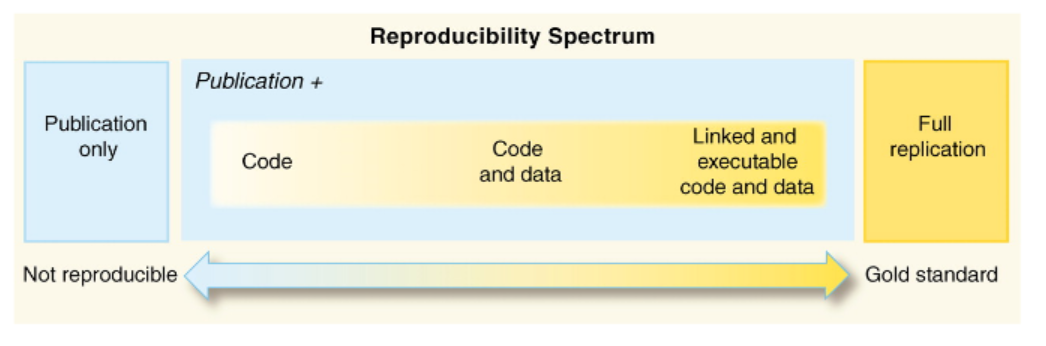
[4]
En pratique, que faire, quels outils utiliser ? (1/2)
Aller de façon pragmatique vers une documentation accrue de ce que l’on fait (comment, pourquoi) et des données que l’on utilise et produit.
- Rendre accessible ses données à soit, aux partenaires, à tous) :
- Documenter collectivement ses pratiques de gestion de données (responsabilités, formats, cycle de vie, archivage…) dns un Data Management Plan qui doit être vivant et partagé.(Opidor)
- Dépôt internationaux (ENA, NCBI) pour les données specialisées
- DataVerse, Figshare, Zenodo ou autre pour les autres données
- Définir et fixer les versions des outils utilisés :
- Conda, Bioconda
- Singularity, Docker
- Machine Virtuelle
En pratique, que faire, quels outils utiliser ? (2/2)
- Décrire son workflow d’analyse, le rendre portable :
- Galaxy
- Snakemake, Nextflow
- Gérer les versions de ses codes, les publier :
- git
- GitHub / GitLab
- Tracer son analyse dans des documents computationnels partageables et réutilisables :
- Rmd
- Jupyter Notebooks
Objectifs du TP
Décomplexifier les problème, se décomplexifier sur ses pratiques , désacraliser la reproductibilité !
Vous fournir des outils, des pistes pour rendre vos projets :
- transparents
- robustes
- réutilisables
- partageables
Bref, plus ouverts et reproductibles.
Organiser son espace de travail
Organiser son espace de travail
[5]
Séparer :
- données
- code
- scripts
- résultats
- Avoir un copie de sauvegarde de ses données
- Mettre le répertoire et les fichiers de données en lecture seule
- Avoir une convention de nommage de ses fichiers
Data management tips (Source : Kira Höffler | PDF)

Versionner ses documents
Pourquoi versionner ses projets ?

→ Suivre l’évolution des fichiers
- garder en mémoire chaque modification de chaque fichier
- pourquoi elle a eu lieu
- quand et par qui !
→ Faciliter le développement collaboratif
- fusionne les différentes modifications
→ Revenir à une version précédente
- et assure une sauvegarde de son travail.
Vocabulaire
- Repository = Dépôt. Dossier contenant tous les fichiers d’un projet. Personnel ou partagé. Public ou privé. Local ou distant (remote).
- Commit = Enregistrement d’un ensemble de fichier à un instant T (= photo)
- Branche = Ensemble chaîné de commits, par défaut la branche principale s’appelle « main »
- Git : logiciel open-source de gestion de version de document. Il est tout a fait possible d’utiliser git pour versionner ses documents sans GitHub.
- GitHub : site web permettant de centraliser en ligne ses dépôts git et facilitant la collaboration sur les projets.

Travailler en commun avec Git et Github :
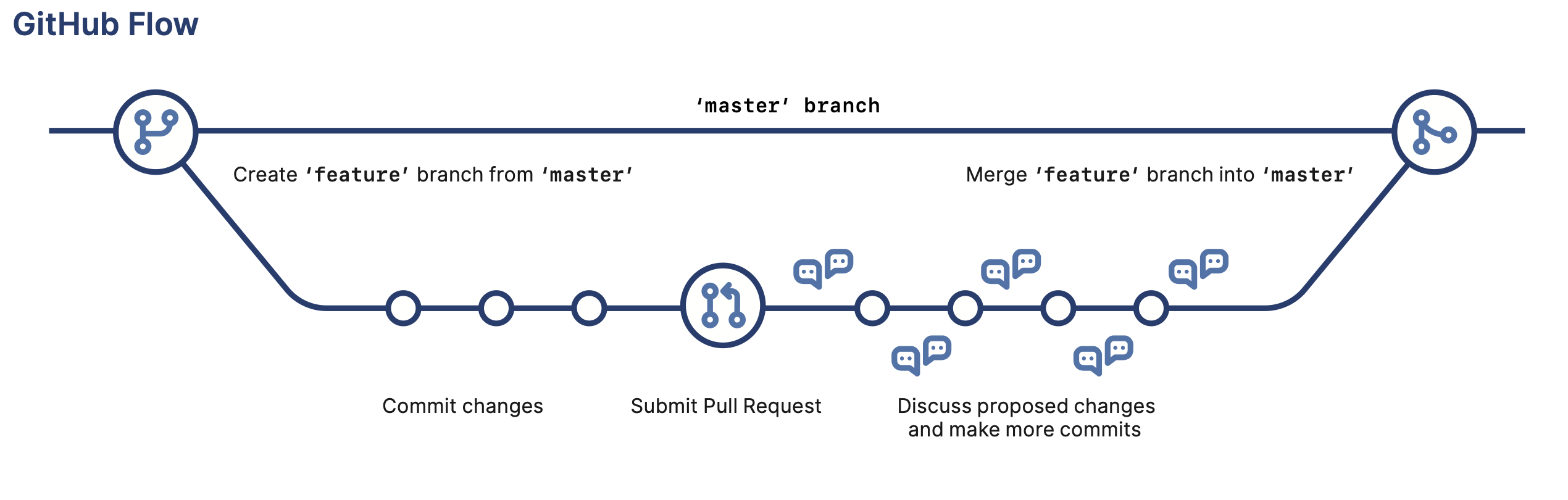
- Branche = version parallèle à la version principale
- Pull Request = demande de fusion des modifications d’une branche vers la branche principale
TP : Utilisation de Git et GitHub
Git : en ligne de commandes
git clone: cloner un dépot distantgit init: initialiser le versionning sur un dépôt localgit commit: enregistrer l’état d’un dépôtgit status: afficher l’état des documents du dépôtgit diff: comparer l’état actuel au dernier commit, ou deux commits entre eux ou deux branchesgit pull: récupérer les commits distantsgit push: envoyer les commits locaux
et bien d’autres encore (blame, revert,…)
Des langages à faible balisage pour faciliter la traçabilité et la prise de note
Markdown - exemples de mise en forme
# Titre H1
## Sous-titre H2
### Sous-titre H3
*italique*, **gras** et `code`
> Citations
```
bloc de code
```
* liste
* item
* item
[lien](https://fr.wikipedia.org)

#lien vers une image en ligne ou dans l'espace de travailGardez ce mémo à porté de main !
Exemple illustrant la simplification du balisage
HTML
<ul>
<li>item1</li>
<li>item2</li>
</ul>Markdown
- item1
- item2Documents computationels - Notebook
Documents computationels - Notebook
- Il faut se donner les moyens pour qu’autrui puisse inspecter nos analyses
- Expliciter pour augmenter les chances de trouver les erreurs et de les éliminer
- Inspecter pour justifier et comprendre
- Refaire pour vérifier, corriger et réutiliser

Documents computationels - Notebook
Regrouper dans un unique document: les informations, le code, calculs et les résultats ; pour assurer leur cohérence et améliorer la traçabilité. Tout en étant exportable (ex : html) pour une meilleure portabilité et lisibilité.
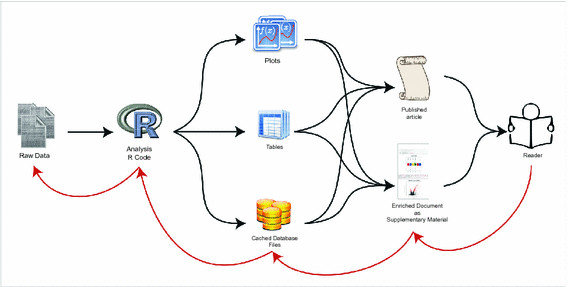
Encore un joli mémo pour R markdown.
D’un RMarkdown à un PDF

 ]
]
Nouveautés : Quarto
Une alternative open-source pour la publication de documents scientifiques et techniques !
- Input :
Python,R,Julia, etObservable - Output : Articles, présentations, livres, sites/blogs, …
- Rédaction et déploiement simplifié

TP : Documents computationels
Documents computationels : En résumé
- Dans un unique document on a :
- Une entête générale
- Du texte, mis en forme avec markdown
- Du code R ou Python (ou autre) dans des chunks
- Des résultats, plots et outputs
Ce document est versionné, dans un format texte dont la lisibilité est assurée au cours du temps, exportable en HTML et accessible via les GitHub Pages.
→ Expliciter pour trouver les erreurs et de les éliminer
- Inspecter pour justifier et comprendre
- Refaire pour vérifier, corriger et réutiliser
Aller vers le FAIR et rédiger son PGD
Science Ouverte
Second Plan Nation Science Ouverte (2022)
La science ouverte est la diffusion sans entrave des publications et des données de la recherche.
Elle s’appuie sur l’opportunité que représente la mutation numérique pour développer l’accès ouvert aux publications et –autant que possible– aux données de la recherche.

Exigences des financeurs
Open Access :
- Obligation to deposit Pre-print in Open repositories (HAL or equivalent)
- Favor Gold (at least) Open Access Journals
- Favor Creative Commons “CC-BY” license
Open Research Data :
- Alignement on European and National strategy
- « As open As Possible, as Closed as Necessary »
Plan de Gestion de Données - Quoi ?
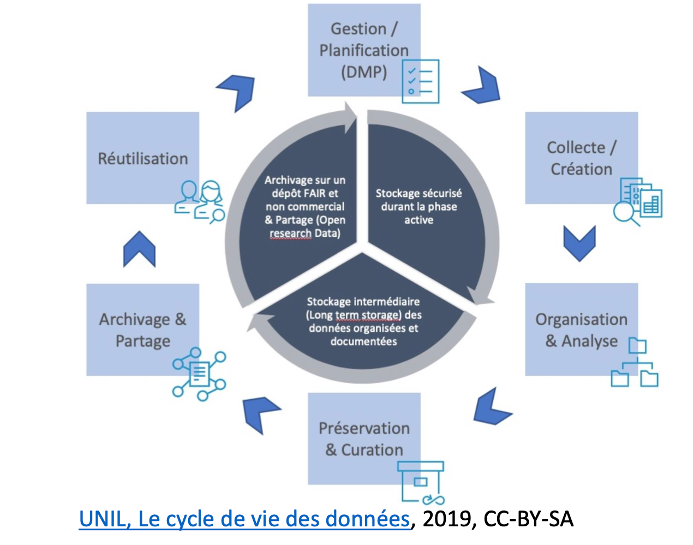
Un document collaboratif qui définit la façon dont les données seront gérées pendant et après le projet.
Objectifs
Penser à toutes les étapes du cycle de vie de la donnée
Avantages
Le modèle vous aide à anticiper l’ensemble des questions et des problèmes qui peuvent se poser par le biais d’une série de questions.
Plan de Gestion de Données - Quand ? Qui ?
Quand ?
Document évolutif. Au moins 3 versions :
- Une première version au début du projet
- Des mises à jour régulières tout au long du projet
- Une version finale en fin de projet
Qui
l’ensemble des partenaires. PGD = pour générer du dialogue.
Plan de Gestion de Données - Pourquoi ?
Plan :
On planifie , on anticipe
Gestion :
On gère, on améliore et on commence par ne plus perdre de données.
Données
Quelles sont les données critiques généres ou utilisées dans le projet ?
Objectifs du PGD
- Assurer la reproductibilité des expériences
- Décrire comment les données sont obtenues
- Faciliter la réutilisation des données
- Assurer la bonne compréhension des données
- Respecter la loi et les individus
- Clarifier le cadre légal et éthique
- Éviter la perte de données
- Assurer un stockage approprié (à court et à long terme)
- Clarifier les droits de réutilisation
- Préciser les conditions de partage
- Établir le rôle de chaque personne
- Définir les responsabilités
Objectifs du PGD
Pour vous et vos collaborateurs :
- Se référer au DMP pour :
- Récupérer des données
- Comprendre les données
- Savoir où se trouvent les données
Pour la communauté scientifique, publiez le DMP pour indiquer :
- Quelles données ont été générées dans le cadre du projet
- Où elles sont publiées
- Qui peut y accéder, sous quelles conditions…
PGD en pratique
Différents modèles (ANR, Horizon Europe) - Un seul document avec 6 sections, 2 à 4 questions par section. - DMP OPIDoR pour organiser l’écriture collaborative
DMP Section
Modèle ANR
Informations générales sur le projet
- Financement, date
- Partenaires
- Contributeurs
- Nom, contact et rôles
- Produits de recherche du projet
Produit de recherche ?
Selon l’OCDE, les données scientifiques (research data) sont « des enregistrements factuels (chiffres, textes, images et sons), qui sont utilisés comme sources principales pour la recherche scientifique et sont généralement reconnus par la communauté scientifique comme nécessaires pour valider des résultats de recherche. »
Contenu du DMP
- Informations générales
- Description des données
- Documentation et qualité des données
- Stockage et sauvegarde pendant le processus de recherche
- Exigences légales et éthiques
- Partage des données et conservation à long terme
- Responsabilités et ressources en matière de gestion des données.
Explorons un exemple de DMP
Pour aller + loin
Fixer et partager son environnement
- Conda et Bioconda
- gestion des dépendances, versions
- Possibilité de créer un environnement par analyse
- Exporter son environnement dans un fichier
env.ymlet le versionner conda env export > environment.yml
- Containers, machines virtuelles
- Docker, Singularity, VM virtualbox
- Pour les outils non “conda-isables”, les environnements complexes
- Les images Singularity sont déployables sur les infrastructures type IFB et s’éxecutent “presque” comme un executable
Fixer et partager son environnement (2)

[7]
Gestionnaires de workflows
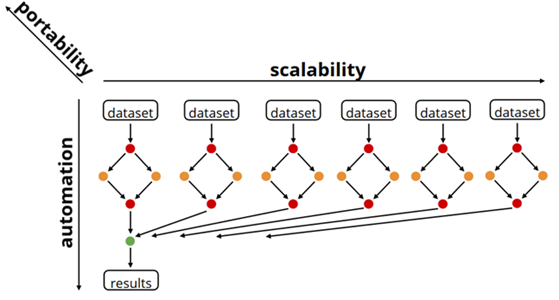
Snakemake ou Nextflow pour définir de façon “simple” et modulaire des workflows d’analyse :
- Parallélisables : les étapes indépendantes peuvent être jouées en parallèle.
- Qui assurent la reprise sur erreur : si on refait une analyse, change un paramètre, seul ce qui doit être rejoué est relancé.
- Portables : un même script peut être joué en local, sur des clusters différents en changeant le fichier de configuration.
- Partageables : un fichier texte versionné.
- Peut gérer pour vous le versionning et l’installation des outils avec conda
exemple de Snakefile
Bash
for sample in `ls *.fastq.gz` do
fastqc ${sample}
doneSnakefile
SAMPLES = glob_wildcards("./{sample}.fastq.gz")
rule final:
input:expand("fastqc/{sample}/{sample}_fastqc.zip", sample=SAMPLES)
rule fastqc:
input: "{sample}.fastq.gz"
output: "fastqc/{sample}/{sample}_fastqc.zip"
conda: "fastqc.yaml"
message: """Quality check"""
shell: """fastqc {input} --outdir fastqc/{wildcards.sample}"""Pour aller + loin - FAIRifier ses données
Dépôts dans les dépôts publics :
- Dans les dépôts thématiques internationaux (européens !)
- données brutes
- données analysées
- Attention aux méta-données
- dans les dépots généralistes (dataverse , figshare, …)
- fichiers tabulés; “autres” données. ce qu’on mettrait en suypplementary material.
- (éventuels) liens vers les fichiers de données
Publier un data-paper ? et un protocol ?
Pour aller + loin - Mat & Met
jusqu’où aller dans la reproductibilité ?
- Mat et Met électroniques :
- Galaxy Pages
- Gigascience, GigaDB :
- “GigaScience aims to revolutionize publishing by promoting reproducibility of analyses and data dissemination, organization, understanding, and use.”
Au final, toujours se poser la question du rapport coût / bénéfice.
Épilogue
Ressources
- FUN MOOC Recherche Reproductible
- FAIR Bioinfo
- Cours Git et Github
- Github pages
- Rmd the definitive Guide
- Snakemake
- NextFlow et nf-core
- Les mémo présentés dans ce cours :
Ressources
Sources

Module 25



Comment mettre en forme et structurer simplement un document texte ?
→ avec un balisage faible tel que Markdown