Objectifs pédagogiques
A l’issue de la formation, les stagiaires connaîtront les principales fonctionnalités du logiciel PyMOL. Ils seront capables de les appliquer pour visualiser leur système biologique d’intérêt, et d’effectuer des commandes basiques d’identification de poches catalytiques, de profilage de surface électrostatique, et de mutations d’acides aminés.
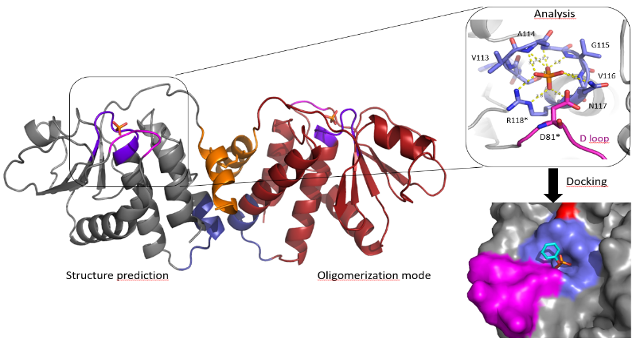
Aussi, ils connaîtront les bases et les outils de bioinformatique structurale et seront autonomes pour effectuer des modèles de protéines par prédiction (Alphafold2), calculer les meilleures poses de fixation de leur(s) ligand(s) (Autodock4) et reconstruire l’éventuel assemblage biologique.
Bonus : Ils s’approprieront ces outils avec une demi-journée dédiée à la modélisation de leur système d’étude : protéines, interactions protéines/ADN, arrimage de ligand, etc.
Programme
Visualiser :
- Maîtriser les bases de la visualisation des protéines en 3D avec PyMOL.
Comprendre :
Analyser des structures 3D de protéines (RX ou RMN).
Identifier des homologues avec HHpred.
Modéliser par prédiction sa protéine d’intérêt avec Alphafold2.
Prédire :
️
- Points forts et limites des différents outils
- ️“hand- on tutorials”
- Plus une session dédiée : «bring your own protein»
Intervenants
Gwenaëlle André / Sylvain Marthey
A work by Migale Bioinformatics Facility
Université Paris-Saclay, INRAE, MaIAGE, 78350, Jouy-en-Josas, France
Université Paris-Saclay, INRAE, BioinfOmics, MIGALE bioinformatics facility, 78350, Jouy-en-Josas, France