git clone git@forge.inrae.fr:migale/quarto.gitDocumentation interne
Le site web déployé
https://documents.migale.inrae.fr est le portail d’accès aux documents générés par les membres de la plateforme Migale.
| URL | Statut | Description |
|---|---|---|
| https://documents.migale.inrae.fr | Page d’accueil | |
| https://documents.migale.inrae.fr/training-materials.html | Liste des supports de formation | |
| https://documents.migale.inrae.fr/news.html | Liste des newsletters | |
| https://documents.migale.inrae.fr/presentations.html | Liste des présentations | |
| https://documents.migale.inrae.fr/analysis_report_listing.html | Liste des rapports d’analyse | |
| https://documents.migale.inrae.fr/intranet.html | Liste des documents internes |
Présentation du dépôt
Le dépôt est accessible sur la forge INRAE pour les membres du groupe migale. Pour cloner le dépôt :
Il n’est pas indispensable d’installer quarto sur sa machine en local, mais c’est préférable pour plusieurs raisons :
- pour visualiser le rendu
- tester que la page compile correctement
- pour éviter que la page soit compilée par quarto au moment du déploiement du site sur le runner permettant le déploiement automatique (le contenu du répertoire
_freezen’est pas recompilé)
Organisation des documents
Les documents sont rédigés au format qmd. Ils peuvent être :
- des documents HTML
- des dashboards
- des présentations
Ajouter un rapport d’analyse
Préparer le dossier du projet
Les rapports d’analyse sont stockés dans le répertoire posts/analyses/. Chaque rapport a son propre répertoire, qu’il faut anonymiser un minimum en rajoutant 5 chiffres après le nom du projet (ex: bidou-12345)
Organisation du dossier
4 fichiers sont indispensables à la racine du projet :
- le fichier
index.qmdest le document principal qui contient les analyses effectuées - le fichier
follow-up.qmdest un lien symbolique vers le fichierposts/analyses/template/follow-up.qmd - le fichier
infos.tsv - le fichier
preview.pngest l’image du projet. Elle est visible sur les pages qui listent les projets et dans le fichier de suivi du projet.
Les répertoires suivants permettent d’organiser le contenu des autres fichiers :
- un répertoire
datapermet de stocker les fichier de données liés au projet - un répertoire
htmlpermet de stocker les fichiers HTML liés au projet (output d’outils par exemple) - un répertoire
docspermet de stocker les documents importants liés au projet - un répertoire
imagesouimgpermet de stocker les images externes affichées dans le rapport - un répertoire
scriptpermet de stocker les scripts développés dans le projet
Un exemple d’organisation et de fichiers est disponible dans le répertoire posts/analyses/template/.
- Le fichier
infos.tsvdoit être rempli à chaque étape du projet- réception de la demande
- deadline demandée
- changement de deadline
- réunions
- sauvegarde des données
- réception du questionnaire de satisfaction
- soumission d’un papier
- tout ce qui vous semble pertinent…
Ce fichier est lu par le fichier follow-up.qmd au moment de sa compilation et génèrera la fiche de suivi du projet.
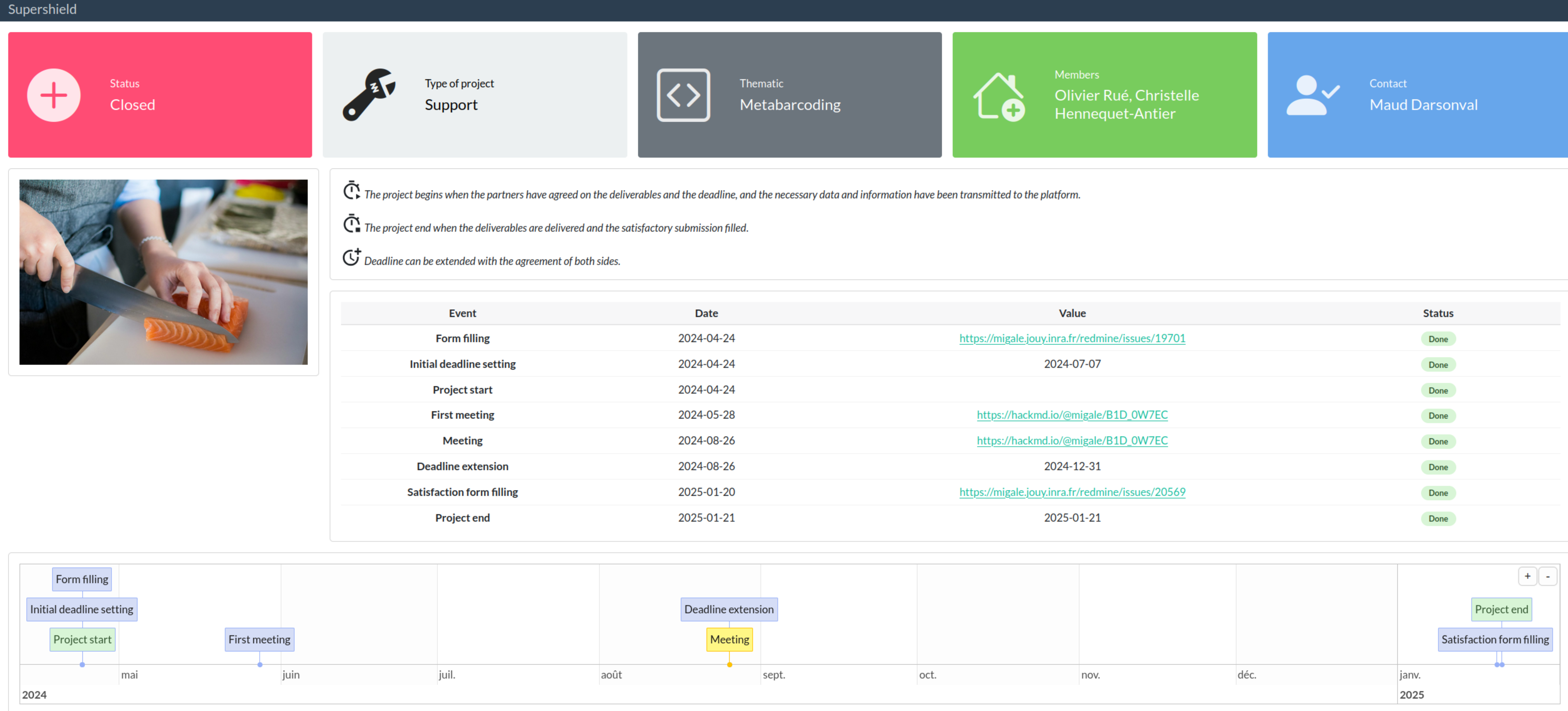
Renseigner les informations générales du projet dans le fichier resources/projects.tsv
Les informations générales concernant le projet (demandeur, mots-clés…) sont à renseigner dans le fichier resources/projects.tsv. C’est à partir de ce fichier que sont créés les documents de synthèse à propos des projets (project_metrics.qmd et data-analysis.qmd)
Voici les informations demandées :
Name | Type | Analysis | Status | City | Organization | Organization details | Department | Team | Contact | Begin | End | Valorisation | ValorisationDate | Report | Migale contact | Repository | Bioproject | Cleanup | ContactPosition | PhDGuidance | Key-words |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
NEBULA | Collaboration | Metabarcoding | Closed | Montpellier | INRAE | INRA | PHASE | UMR DMEM | Bénédicte Goustard | 2023-11-20 | 2024-12-02 | nebula-29873 | Olivier Rué,Mahendra Mariadassou | /backup/partage/migale/NEBULA/RAW_DATA/ | Researcher | No | 16S ; metabarcoding ; illumina ; mice ; physical activity |
| Colonne | Signification | Valeur |
|---|---|---|
| Name | Nom du projet | string |
| Type | Type du projet | Collaboration, Support |
| Analysis | Thématique | MicroArray, Metabarcoding, Genomics, Metagenomics, Metatranscriptomics, Transcriptomics, Genome assembly, Text-mining |
| Status | Statut du projet | Rejected, Closed, In progress, To come |
| City | Ville du demandeur | string |
| Organization | Institut | Private, Université, INRAE, ENVA, CNRS, MNHN, INSERM, Université Paris-Saclay, UTC, CHRU |
| Organization details | Nom de la societé privée, nom de l’université… pas nécessaire pour les unités INRAE | string |
| Department | Département pour les unités INRAE | MICA, SA, EA, GA, PHASE, HBAN, SPE, BAP, MathNum, AQUA |
| Team | Nom de l’équipe ou de l’unité | string |
| Contact | Prénom et nom du ou des demandeurs | string, séparés par une virgule |
| Begin | Date de début du projet | Date (format YYYY-MM-DD) |
| End | Date de fin du projet | Date (format YYYY-MM-DD) |
| Valorisation | Lien vers la ou les valorisations (DOI), séparées par une virgule | URL |
| ValorisationDate | Date de la valorisation | Date (format YYYY-MM-DD) |
| Report | Nom du répertoire du projet | string |
| Migale contact | Prénom et nom de la ou des personnes en charge du projet côté Migale | string, séparés par une virgule |
| Repository | Path contenant une copie des données essentielles le temps de l’analyse | /path/vers/le/repertoire/ |
| Bioproject | Identifiant du BioProject sur les dépôts publics | string (PRJXXXXX) |
| Cleanup | Statut de la conservation des données sur notre infrastructure | NA, REMOVED, /work_projet/genorhizo/, WAITING, NO |
| ContactPosition | Statut du demandeur | NA, PhD student, Researcher, Engineer, Technician, Postdoc |
| PhDGuidance | Encadrement d’un thésard pendant le projet ? | NA, Yes, No |
| Key-words | Mots-clés du projet | string séparés par des ; |
Fin de projet
Une fois le projet terminé, il faut déplacer le répertoire dans un autre dépôt : https://forgemia.inra.fr/migale/backup-data-analysis-reports. Il faut alors remplacer son contenu par :
- un fichier
index.htmlcontenant la redirection vers le nouveau dépôt
<html>
<meta http-equiv="refresh" content="0;url=https://backup-data-analysis-reports-6cf7b2.pages.mia.inra.fr/posts/analyses/holovini-pilote-76384/">
</html>- un fichier
follow-up.html
<html>
<meta http-equiv="refresh" content="0;url=https://backup-data-analysis-reports-6cf7b2.pages.mia.inra.fr/posts/analyses/holovini-pilote-76384/follow-up.html">
</html>Ajouter une news
Ajouter un tutoriel
Ajouter des supports de formation
Pense-bête quarto
Référence à un outil
Pour faire référence à un outil, il faut ajouter une entrée au format bibtex dans le fichier /resources/biblio.bib. Ensuite on peut y faire référence dans les documents avec la syntaxe @toolname (toolname étant l’identifiant de l’entrée bibtex). Exemple:
FROGS @frogs is...FROGS [1] is…
The reference is also added in the References section at teh bottom of the document.
Description d’un outil
Il est possible d’utiliser la bibliothèque iconify pour mettre en avant les outils/packages/banques dont on parle dans les documents. Par exemple:
- seqkit
[2]
**seqkit** {{< iconify mdi tools >}} @seqkit- Silva version 138.1
[3]
**Silva version 138.1** {{< iconify mdi database >}} @silva- phyloseq
[4]
**phyloseq** {{< iconify mdi package-variant >}} @phyloseqLa liste des icônes est disponible ici : https://icon-sets.iconify.design/
Boutons téléchargement
Il est possible avec l’extension downloadthis d’ajouter facilement des boutons pour le téléchargement d’un fichier. Exemple :
{{< downloadthis files/quarto.png >}}Full documentation here: https://shafayetshafee.github.io/downloadthis/example.html
Includes
Il est possible d’insérer du texte venant d’un autre fichier .qmd dans un fichier .qmd. Pour cela on utilise les includes, avec la syntaxe suivante :
::: callout-note
This document is a report of the analyses performed. You will find all the code used to analyze these data. The version of the tools (maybe in code chunks) and their references are indicated, for questions of reproducibility.
:::C’est très pratique pour des blocs génériques comme l’introduction ajoutée au début de chaque rapport d’analyse:
This document is a report of the analyses performed. You will find all the code used to analyze these data. The version of the tools (maybe in code chunks) and their references are indicated, for questions of reproducibility.
Sécurisation des rapports
Pour les rapports qui n’ont pas vocation à être accessibles, il est possible de les protéger par mot de passe avec l’outil staticrypt.
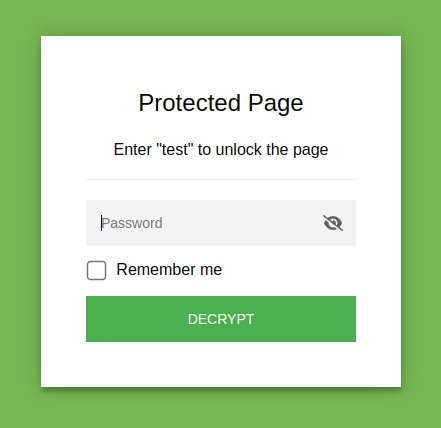
Staticrypt est un outil qui permet de protéger par mot de passe une page HTML statique en la chiffrant avec AES-256 dans le navigateur.
- Aucun backend requis : tout fonctionne côté client.
- On génère un fichier HTML chiffré à partir d’un fichier source.
- Un mot de passe saisi par l’utilisateur déchiffre et affiche la page.
2 étapes sont nécessaires pour mettre en place cette sécurisation :
- Créer une variable dans le menu CI/CD de Gitlab Pages qui contiendra le mot de passe
- Créer une ligne dans le fichier
.gitlab-ci.yamlpour lancerstaticryptaprès le render du site
...
- quarto render
- staticrypt .public/posts/datavzrd/index.html -d .public/posts/datavzrd/ -p $TEST_PASSWD --template-color-primary "#fd904e" --template-color-secondary "#5f999d" --template-title "Protected Report"
- mv .public public
...Dans cet exemple c’est le rapport datavzrd qui est protégé par le contenu de la variable $TEST_PASSWD
