library(cluster)Initiation à R
Introduction
Ce post a pour objectif de vous aider à débuter sous R : les structures de données et leur manipulation, les principales fonctionnalités du langage et de quoi faire des figures très simples. Ce tutoriel est adapté du document rédigé par Schbath&Schaeffer (2010) comme support à leur formation Initiation au langage R de la plateforme Migale.
Historique
R est un logiciel libre basé sur le langage de programmation S, langage inventé chez AT & T Bell Laboratories par John Chambers et ses collègues (Becker et al., 1988). L’approche S est vite devenue très populaire dans les milieux académiques du fait de son caractère interactif et flexible. En effet, les utilisateurs peuvent implémenter leurs propres techniques et les diffuser sous forme de bibliothèques. Ainsi, il existe un très grand nombre de bibliothèques, disponibles sur Statlib, un système de distribution de logiciels statistiques. A l’origine, S n’existait pas sous MacIntosh. Or Robert Gentleman et Ross Ihaka à l’Université d’Auckland en Nouvelle Zélande, étaient équipés de Mac. Ils décidèrent alors, au début des années 1990, d’initier un projet pour fournir un ”environnement statistique” à leur laboratoire. Comme ils connaissaient S, ils choisirent d’implémenter une syntaxe à la S, et déposèrent leur logiciel sur Statlib. Puis en 1995, R est devenu un logiciel libre, imposant que toute modification reste dans le domaine du libre. R s’est depuis, largement développé, sans doute du fait d’Internet qui a permis à des personnes géographiquement dispersées de collaborer et d’y apporter des améliorations.
Environnement
R est distribué librement sous les termes de la GNU, General Public Licence ; son développement et sa distribution sont assurés par plusieurs statisticiens rassemblés dans le R Development Core Team (http://www.r-project.org/). R est écrit principalement en C et parfois en Fortran. Pour les machines Windows, Linux et Macintosh, R est disponible sous forme d’exécutables précompilés. Pour télécharger une version du logiciel, il faut passer par CRAN (Comprehensive R Archive Network ), un réseau mondial de sites miroirs qui stockent de façon identique les différentes versions, les mises à jour, les bibliothèques et la documentation. Afin d’optimiser le chargement, on choisit généralement le site miroir le plus près de chez soi. En France, il existe plusieurs sites dont la liste est ici. Une fois R installé, il suffit de lancer le programme correspondant (typiquement en tapant R sous Unix/Linux). Deux fichiers .Rhistory et .RData sont alors automatiquement créés dans le répertoire courant, et le symbole > apparaît indiquant que R est prêt.
Comme on va le voir, R travaille avec des données et des fonctions qui permettent de manipuler ces données. Plusieurs bibliothèques de fonctions sont installées par défaut (base, graphics, stats, etc. . .). Pour utiliser des bibliothèques spécifiques, à condition que celles-ci aient été chargées, il est nécessaire de les appeler en utilisant la commande library. Par exemple, les fonctions de classification développées par Rousseeuw et ses collaborateurs sont contenues dans la bibliothèque cluster. Pour les utiliser, il faut donc appeler cette bibliothèque :
Si la bibliothèque appelée n’est pas chargée sur votre machine, R retourne un message d’erreur. Par exemple, si on appelle la la bibliothèque “actuar” qui n’est pas chargée, on obtient :
library(actuar)
Attachement du package : 'actuar'Les objets suivants sont masqués depuis 'package:stats':
sd, varL'objet suivant est masqué depuis 'package:grDevices':
cmLa liste de toutes les bibliothèques existantes est accessible sur le site du CRAN sous la rubrique packages.
Quelques fonctions utiles
getwd()permet de connaître le répertoire dans lequel on travaille.dir()permet de connaître le contenu du répertoire de travail.setwd("repertoire")permet de changer de répertoire de travail.source("script.r")exécute un fichier script.save(monobjet,file="monobjet.rdata")permet de sauver l’objet monobjet sous le format R. Il est aussi possible de sauver plusieurs objets dans le même fichier. Par exemple :save(objet1,objet2,file="mesobjets.rdata").load("mesobjets.rdata")permet de recharger des objets sauvegardés au cours d’une session précédente.write.table(montableau,file="montableau.txt")permet de sauvegarder le tableau de données montableau sous un format .txt.
Noter que l’appel à une fonction se fait toujours sous la forme fonction(arguments, options) et que les paranthèses sont indispensables même sans arguments ni options.
Pour quitter R, il faut utiliser la commande q(). R pose alors la question :
Save workspace image? [y/n/c]:Si vous répondez y, R sauvegarde le fichier .RData, qui contient tous les objets créés au cours de la session. Si vous répondez n, ces objets sont perdus. Pour continuer la session, il faut répondre par la lettre c. L’historique des commandes est conservé automatiquement dans le fichier .Rhistory.
Le système d’aide
Il existe une aide directement disponible dans R : on y accède en utilisant le ? suivi du nom de la fonction, ou bien help(nom.fonction). Si la fonction appartient à une bibliothèque particulière, il faut que celle-ci soit chargée. En réponse à cette commande d’aide, le logiciel affiche une page comportant :
- le nom de la fonction et le nom de la bibliothèque à laquelle elle appartient,
- le titre de la fonction,
- une brève description de la fonction et de son utilisation,
- une description détaillée des arguments de la fonction, et du type d’objet qu’elle retourne,
- des références, des liens vers des fonctions similaires et des exemples.
Nous pouvons, par exemple, obtenir la documentation de la fonction “mean” avec la commande :
help(mean)mean package:base R Documentation
Arithmetic Mean
Description:
Generic function for the (trimmed) arithmetic mean.
Usage:
mean(x, ...)
## Default S3 method:
mean(x, trim = 0, na.rm = FALSE, ...)
Arguments:
x: An R object. Currently there are methods for numeric data
frames, numeric vectors and dates. A complex vector is
allowed for ' trim = 0 ' , only.
trim: the fraction (0 to 0.5) of observations to be trimmed from
each end of ' x ' before the mean is computed.
na.rm: a logical value indicating whether ' NA ' values should be
stripped before the computation proceeds.
...: further arguments passed to or from other methods.
Value:
For a data frame, a named vector with the appropriate method being
applied column by column.
If ' trim ' is zero (the default), the arithmetic mean of the values
5in ' x ' is computed, as a numeric or complex vector of length one.
If any argument is not logical (coerced to numeric), integer,
numeric or complex, ' NA ' is returned, with a warning.
If ' trim ' is non-zero, a symmetrically trimmed mean is computed
with a fraction of ' trim ' observations deleted from each end
before the mean is computed.
References:
Becker, R. A., Chambers, J. M. and Wilks, A. R. (1988) _The New S
Language_. Wadsworth & Brooks/Cole.
See Also:
'weighted.mean ' , ' mean.POSIXct '
Examples:
x <- c(0:10, 50)
xm <- mean(x)
c(xm, mean(x, trim = 0.10))
mean(USArrests, trim = 0.2)Il existe également une aide au format html permettant l’utilisation d’un moteur de recherche ; on y accède en utilisant help.start() qui ouvre la page d’accueil de l’aide de R, dans la fenêtre d’un navigateur interne. Il existe aussi des commandes permettant une investigation de la documentation à partir de mots-clés :
- la fonction
apropos("keyword")retourne la liste de toutes les fonctions qui contiennent dans leur nom, le mot-clé recherché. - la fonction
help.search("keyword")retourne la liste de toutes les fonctions qui contiennent dans leur nom ou dans leur titre, le mot-clé recherché.
La fonction args donne en général la liste des arguments d’une fonction. Par exemple :
args(sort)function (x, decreasing = FALSE, ...)
NULLUn conseil : ne pas hésiter à consulter l’aide d’une fonction la première fois qu’on l’utilise car on peut y découvrir des options intéressantes.
Rstudio
RStudio est un environnement de développement intégré libre, gratuit, et qui fonctionne sous Windows, Mac OS X et Linux. Il complète R et fournit un éditeur de script avec coloration syntaxique, des fonctionnalités pratiques d’édition et d’exécution du code (comme l’autocomplétion), un affichage simultané du code, de la console R, des fichiers, graphiques et pages d’aide, une gestion des extensions, une intégration avec des systèmes de contrôle de versions comme git, etc. Il intègre de base divers outils comme par exemple la production de rapports au format Rmarkdown. Il est en développement actif et de nouvelles fonctionnalités sont ajoutées régulièrement. Pour une présentation plus générale de RStudio on pourra se référer au site du projet : http://www.rstudio.com/.
RStudio peut tout à fait être utilisé pour découvrir et démarrer avec R.
R est utilisable sur l’infrastructure Migale en tapant R dans la console ou via l’interface de développement rstudio. Un compte vous est nécessaire pour y accéder. Remplissez ce formulaire pour obtenir un compte.
Langage de programmation
R est un langage interprété et non compilé, autrement dit les commandes tapées au clavier sont directement exécutées lorsque l’on appuie sur la touche “Entrée”. On pourra soit taper une commande par ligne, soit taper plusieurs commandes séparées par le symbole ; sur une même ligne. Une commande peut s’écrire sur plusieurs lignes, auquel cas R matérialise le début de la 2ème ligne d’instructions par le symbole +. R est un langage simple et intuitif dans lequel chaque chose est un objet. Utiliser R revient donc à créer et manipuler des objets. On distingue deux types d’objets, les données et les fonctions.
Les données
Les données se caractérisent par un type (scalaire, vecteur, tableau, . . .), un mode (numérique, caractère, logique), une taille, une valeur et un nom.
Caractéristiques d’un objet
Type
Les données manipulées par R peuvent être de différents types :
- vecteur : suite d’éléments (une dimension),
- tableau : tableau à k dimensions,
- matrice : tableau à 2 dimensions,
- facteur : suite d’éléments catégoriels avec les différents niveaux possibles,
- tableau de données : ensemble de vecteurs de même longueur,
- séries temporelles : jeu de données de type séries temporelles (avec fréquence, dates),
- liste : liste d’objets.
Mode
Les éléments stockés dans un objet peuvent être de 4 modes différents :
- numérique,
- caractère (entre guillemets),
- complexe,
- logique (
TRUEouFALSE)
Pour connaître le mode d’un objet x, on utilise la fonction mode :
x<-0
mode(x)[1] "numeric"titre<-"coucou"
mode(titre)[1] "character"Seuls les tableaux de données et les listes peuvent contenir des éléments de modes différents.
Taille
La taille d’un objet est le nombre d’éléments qu’il contient. Elle s’obtient à l’aide de la fonction length :
length(x)[1] 1Nom
Le nom d’un objet doit obligatoirement commencer par une lettre et ne peut comporter que des lettres, des chiffres, des espaces soulignés et des points.
Valeur
Quelque soit le mode, une valeur manquante est représentée par NA. Voici quelques remarques sur les valeurs numériques.
- R représente correctement les valeurs infinies avec
Infet-Inf:
x<-0
1/x[1] Inf- ainsi que les valeurs qui ne sont pas des nombres avec
NaN:
y<-1/0
y-y[1] NaN- La notion exponentielle est utilisée pour les grandes valeurs :
x<- 100 * 5.3e12
x[1] 5.3e+141/x[1] 1.886792e-15Créer des objets
L’opérateur d’assignation <- est une façon implicite de créer un objet ; le mode et le type de l’objet sont en effet automatiquement déterminés.
x<-2
y<-x
y[1] 2Création d’un vecteur
L’opérateur d’assemblage c() est aussi une façon implicite pour créer un vecteur :
x<-c(1,3,5,7,9)
x[1] 1 3 5 7 9length(x)[1] 5y<-c(x,11,13)
y[1] 1 3 5 7 9 11 13La création d’un vecteur pourra aussi être faite via la génération de suites. On peut aussi créer un vecteur de façon explicite en spécifiant simplement son mode et sa longueur ; la valeur des éléments du vecteur sera alors initialisée selon le mode : 0 pour numérique, FALSE pour logique, "" pour caractère.
x<-vector(mode="numeric", length=5)
x[1] 0 0 0 0 0y<-vector(mode="logical", length=4)
y[1] FALSE FALSE FALSE FALSEz<-vector(mode="character", length=3)
z[1] "" "" ""Les éléments du vecteur seront ensuite modifiés en utilisation l’indexation.
Création d’une matrice
Une matrice est créée à l’aide de la fonction matrix. On spécifie le nombre de lignes et le nombre de colonnes via les arguments nrow et ncol, et les valeurs des éléments de la matrice via l’argument (vectoriel) data. Par exemple, pour initialiser une matrice (2 × 3) à 0, on utilisera :
x<-matrix(data=0, nrow=2, ncol=3)
x [,1] [,2] [,3]
[1,] 0 0 0
[2,] 0 0 0Si l’on veut remplir la matrice avec des valeurs différentes, les valeurs fournies par le vecteur data rempliront alors par défaut les colonnes successives :
x<-matrix(data=c(1,2,3,4,5,6), nrow=2, ncol=3)
x [,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6Pour remplir la matrice plutôt par ligne, il suffit de spécifier l’argument byrow=TRUE :
x<-matrix(data=c(1,2,3,4,5,6), nrow=2, ncol=3, byrow=TRUE)
x [,1] [,2] [,3]
[1,] 1 2 3
[2,] 4 5 6On peut aussi rajouter des lignes (fonction rbind) ou des colonnes (fonction cbind) à une matrice existante (voire concaténer des matrices). Voici un exemple :
cbind(x,c(7,7)) [,1] [,2] [,3] [,4]
[1,] 1 2 3 7
[2,] 4 5 6 7rbind(x,c(8,8,8)) [,1] [,2] [,3]
[1,] 1 2 3
[2,] 4 5 6
[3,] 8 8 8cbind(x,x) [,1] [,2] [,3] [,4] [,5] [,6]
[1,] 1 2 3 1 2 3
[2,] 4 5 6 4 5 6On a la possibilité d’attribuer des noms aux lignes et aux colonnes d’une matrice via l’option dimnames (ces noms doivent être de type character ). Pour connaître les dimensions d’une matrice, on peut utiliser séparément les fonctions nrow et ncol, ou alors la fonction dim (la fonction length retourne le nombre d’éléments de la matrice) :
dim(x)[1] 2 3length(x)[1] 6Création d’une table de données
Un tableau de données est une collection de vecteurs de même longueur. On peut créer un tableau de données en utilisant la fonction data.frame. Par exemple :
seq1<-c(0.2,0.3,0.4,0.1)
seq2<-c(0.25,0.35,0.15,0.25)
df<-data.frame(seq1,seq2)
df seq1 seq2
1 0.2 0.25
2 0.3 0.35
3 0.4 0.15
4 0.1 0.25On peut affecter un nom de colonne particulier (différent du nom du vecteur) directement à la création du tableau de données :
toto<-c(0.25,0.25,0.4,0.1)
df<-data.frame(seq1,seq2, seq3=toto)
df seq1 seq2 seq3
1 0.2 0.25 0.25
2 0.3 0.35 0.25
3 0.4 0.15 0.40
4 0.1 0.25 0.10Il est possible de donner des noms aux lignes avec l’option row.names qui doit fournir un vecteur de mode caractère et de longueur égale au nombre de lignes du tableau de données. Par exemple, dans l’exemple ci-dessus le tableau de données correspond aux compositions en nucléotides de deux séquences, on pourrait donc écrire :
seq1<-c(0.2,0.3,0.4,0.1)
seq2<-c(0.25,0.35,0.15,0.25)
df<-data.frame(seq1,seq2,seq3=toto,row.names=c("A","C","G","T"))
df seq1 seq2 seq3
A 0.2 0.25 0.25
C 0.3 0.35 0.25
G 0.4 0.15 0.40
T 0.1 0.25 0.10On verra plus loin que la fonction read.table qui permet de lire des données dans un fichier crée implicitement un objet de type tableau de données. Les fonctions cbind et rbind que nous avons vu pour les matrices s’appliquent aussi pour des tableaux de données. On notera aussi les fonctions ncol et nrow qui renvoient le nombre de colonnes et le nombre de lignes d’un tableau de données (ou d’une matrice).
Création d’une liste
Une liste est une collection d’objets (non nécessairement de même types) ; elle se crée via la fonction list :
seq1<-c(0.2,0.3,0.4,0.1)
L1<-58402
res<-list(L1,seq1)
res[[1]]
[1] 58402
[[2]]
[1] 0.2 0.3 0.4 0.1Le nombre d’objets contenus dans une liste s’obtient en faisant
length(res)[1] 2Pour rallonger une liste d’éléments, on utilise l’opérateur d’assemblage c() :
espece1<-"phage"
c(espece1,res)[[1]]
[1] "phage"
[[2]]
[1] 58402
[[3]]
[1] 0.2 0.3 0.4 0.1Contrairement à data.frame, la fonction list ne conserve pas les noms des objets ; autrement dit, la longueur de la séquence (L1) est le premier élément de la liste, et la composition en nucléotides est le 2ème élément. On peut attribuer des étiquettes différentes plus explicites aux différents éléments d’une liste, ce qui sera très pratique pour y accéder (sans avoir à se souvenir dans quel ordre ils sont rangés dans la liste !). Par exemple :
seq1<-c(0.2,0.3,0.4,0.1)
L1<-58402
res<-list(longueur=L1,composition=seq1)
res$longueur
[1] 58402
$composition
[1] 0.2 0.3 0.4 0.1On verra plus loin que pour accéder aux champs longueur (resp. composition) de la liste res, on utilisera l’opérateur $ : res$longueur (resp. res$composition).
Création d’une chaîne de caractère
Une fonction très utile pour créer une chaîne de caractère, outre l’opérateur d’assignation <-, est paste qui permet de concaténer des chaînes de caractères mais aussi des objets automatiquement convertis en chaîne de caractères. Ceci est particulièrement utile par exemple lorsque l’on veut créer un fichier de sortie dont le nom est “dynamique”. Voici quelques exemples :
nom<-paste("filename",".","txt",sep="")
nom[1] "filename.txt"prefix<-"resultats."
i<-5
nom<-paste(prefix,i,".ps",sep="")
nom[1] "resultats.5.ps"Par défaut sep=" " et indique le séparateur de concaténation.
Convertir un objet
On peut vouloir convertir un objet d’un type à un autre. Voici quelques exemples :
- numérique → caractère
i[1] 5as.character(i)[1] "5"- numérique → logique
x<-0:3
as.logical(x)[1] FALSE TRUE TRUE TRUE- logique → numérique
ok<-c(TRUE,FALSE,FALSE,TRUE)
as.numeric(ok)[1] 1 0 0 1Lister et supprimer des objets en mémoire
Quand R est utilisé, les variables, les objets, les fonctions, … sont stockées dans la mémoire vive de l’ordinateur. On peut afficher la liste de tous les objets en mémoire en tapant la commande :
ls() [1] "df" "espece1" "i" "L1" "nom" "ok" "prefix"
[8] "res" "seq1" "seq2" "titre" "toto" "x" "y"
[15] "z" Si l’on souhaite se restreindre aux objets dont le nom contient une chaîne de caractère, il suffit de spécifier celle-ci via l’option pattern. Par exemple, si l’on veut connaître la liste des objets commençant par la lettre s, on tape :
ls(pattern="^s")[1] "seq1" "seq2"La commande ls.str() va quant à elle lister tous les objets en mémoire mais aussi en donner leur détail (type, mode, valeur, etc.).
ls.str()df : 'data.frame': 4 obs. of 3 variables:
$ seq1: num 0.2 0.3 0.4 0.1
$ seq2: num 0.25 0.35 0.15 0.25
$ seq3: num 0.25 0.25 0.4 0.1
espece1 : chr "phage"
i : num 5
L1 : num 58402
nom : chr "resultats.5.ps"
ok : logi [1:4] TRUE FALSE FALSE TRUE
prefix : chr "resultats."
res : List of 2
$ longueur : num 58402
$ composition: num [1:4] 0.2 0.3 0.4 0.1
seq1 : num [1:4] 0.2 0.3 0.4 0.1
seq2 : num [1:4] 0.25 0.35 0.15 0.25
titre : chr "coucou"
toto : num [1:4] 0.25 0.25 0.4 0.1
x : int [1:4] 0 1 2 3
y : logi [1:4] FALSE FALSE FALSE FALSE
z : chr [1:3] "" "" ""Pour effacer des objets de la mémoire, on utilise la fonction rm :
rm(x)Si l’on veut maintenant accéder à x, le message suivant apparaît :
xError: objet 'x' introuvablePour effacer tous les objets de la mémoire, on tape rm(list=ls()).
Générer des données
Suites régulières
Une suite d’entiers consécutifs peut s’obtenir avec l’opérateur : ; par exemple :
5:12[1] 5 6 7 8 9 10 11 12Un vecteur est ainsi crée. Cet opérateur est prioritaire sur les opérations arithmétiques :
n<-10
1:(n-2)[1] 1 2 3 4 5 6 7 81:n-2 [1] -1 0 1 2 3 4 5 6 7 8La fonction rep crée un vecteur dont les éléments sont identiques ou répétés :
rep(x=1,times=10) [1] 1 1 1 1 1 1 1 1 1 1rep(x=1:5,times=3) [1] 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5rep(x=1:5,each=2) [1] 1 1 2 2 3 3 4 4 5 5La fonction seq peut générer des suites régulières de nombres de deux manières différentes :
- soit en lui spécifiant, le début, la fin, puis le pas (argument by)
seq(from=2,to=20,by=2) [1] 2 4 6 8 10 12 14 16 18 20- soit en lui spécifiant le début, la fin et le nombre d’éléments (argument
length)
seq(from=1,to=5,length=10) [1] 1.000000 1.444444 1.888889 2.333333 2.777778 3.222222 3.666667 4.111111
[9] 4.555556 5.000000Variables aléatoires
Il est très utile en statistique de pouvoir générer des variables aléatoires selon diverses lois de probabilité. R peut le faire pour un grand nombre de lois via les fonctions de la forme rfunc(n,p1,p2,...) où func indique la loi de probabilité, n est le nombre de variables à générer et p1, p2, … sont les paramètres de la loi. En voici quelques exemples :
| Loi | Fonction R |
|---|---|
| gaussienne | rnorm(n, mean=0, sd=1) |
| uniforme | runif(n, min=0, max=1) |
| Poisson | rpois(n,lambda) |
| exponentielle | rexp(n, rate=1) |
| \(x^{2}\) | rchisq(n, df) |
| binomiale | rbinom(n,size,prob) |
rnorm(10) [1] 0.8922336 0.5614600 -1.1997821 -1.3260487 1.2280064 0.7319931
[7] -0.3426545 0.4475368 -1.2131237 1.0273809runif(10) [1] 0.96834833 0.21904751 0.83229180 0.98472333 0.17048629 0.20732618
[7] 0.12909565 0.98862734 0.38961193 0.06769734rpois(10,lambda=4) [1] 7 7 5 5 1 4 5 4 5 2les fonctions de la forme rfunc ont toutes des petites soeurs de la forme
pfunc(q,p1,p2,...): pour la probabilité cumulée jusqu’àq,qfunc(p,p1,p2,...): pour le quantile associé à la probabilité cumuléep,dfunc(x,p1,p2,...): pour la densité de probabilité enx.
Cela permet ainsi de tracer des densités et de connaî̂tre la probabilité d’être supérieur ou égal à q sous une loi donnée. Si on prend l’exemple de la loi normale de moyenne 0 et de variance 1 avec q = 2.5 (cf. Figure 1), on a :
1-pnorm(2.5)[1] 0.006209665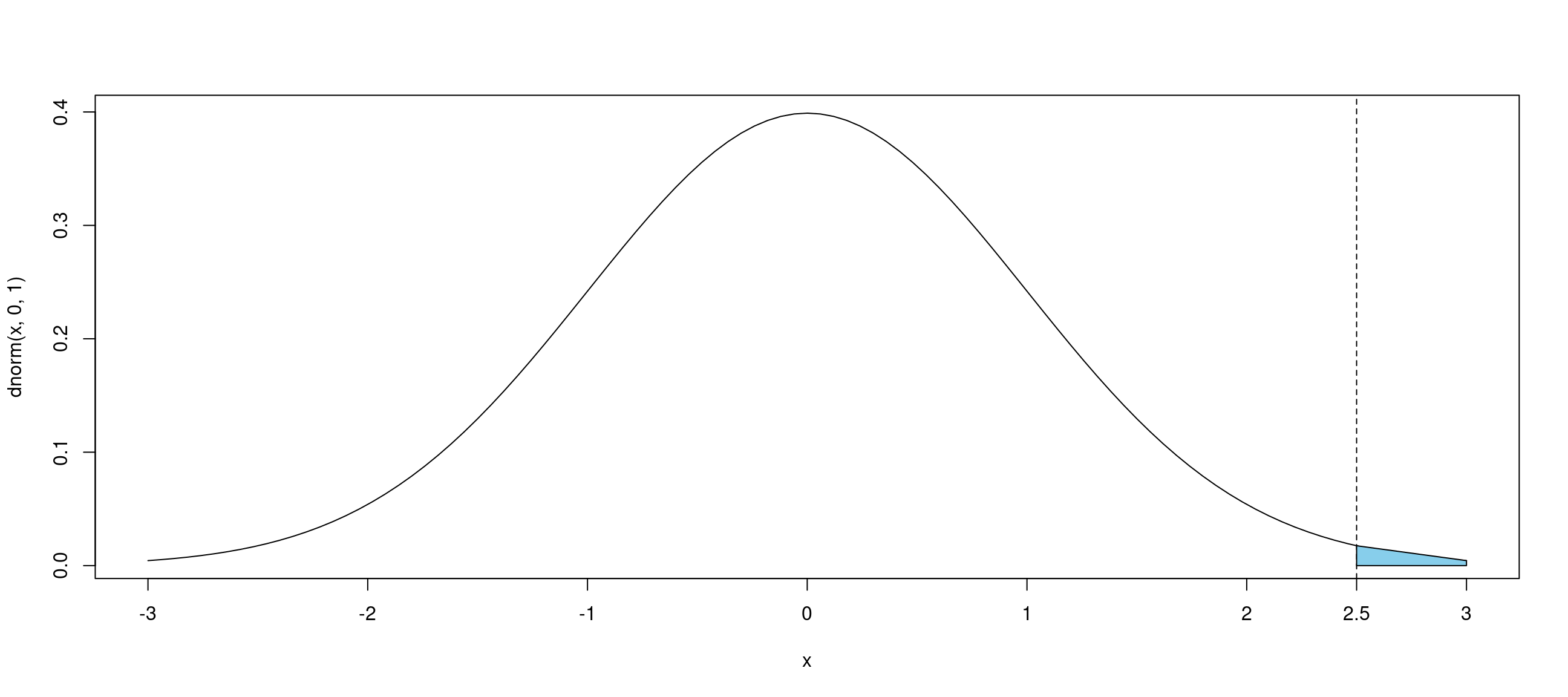
Accéder aux valeurs d’un objet
L’indexation (opérateur []) permet d’accéder de façon sélective aux éléments d’un objet.
Vecteurs
Pour accéder au 2ème élément d’un vecteur x, il suffit de taper x[2]. Cela permet aussi d’aller modifier spécifiquement certains éléments :
x<-1:10
x[2][1] 2x[1]<-10
x [1] 10 2 3 4 5 6 7 8 9 10On peut aussi accéder à plusieurs éléments d’un vecteur et les modifier tous ensemble :
x<- seq(2,20,by=2)
x[1:5][1] 2 4 6 8 10x[1:5]<- rep(0,5)
x [1] 0 0 0 0 0 12 14 16 18 20La commande x[1 :5] est équivalente à x[c(1,2,3,4,5)]. On peut accéder aux éléments dont la valeur vérifie une expression de comparaison. Par exemple, aux valeurs positives d’un vecteur :
x<- runif(10,min=-1,max=1)x>=0 [1] FALSE FALSE FALSE TRUE FALSE FALSE TRUE FALSE FALSE TRUEx[x>=0][1] 0.1270706 0.1497524 0.5657521On peut supprimer un ou plusieurs éléments d’un vecteur en utilisant des indices négatifs :
x<- (1:5)^2
x[-1][1] 4 9 16 25x[c(-2,-4)][1] 1 9 25Matrice
On peut accéder à un élément, à une colonne, à une ligne, à une sous-matrice d’une matrice de la façon suivante :
x<-matrix(data=c(1,2,3,4,5,6), nrow=2, ncol=3)
x [,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6x[1,3][1] 5x[2,][1] 2 4 6x[,3][1] 5 6x[1:2,1:2] [,1] [,2]
[1,] 1 3
[2,] 2 4Noter que x[,3] est un vecteur car, par défaut, R retourne un objet de la plus petite taille. Pour conserver le format d’origine, ici une matrice, il suffit de spécifier l’option drop=FALSE. Par exemple :
x[,3,drop=FALSE] [,1]
[1,] 5
[2,] 6La modification des éléments d’une matrice se fait comme pour les vecteurs. On peut également supprimer une ou plusieurs lignes ou colonnes d’une matrice en utilisant des indices négatifs. Par exemple, pour supprimer la 2ème colonne d’une matrice :
x<-matrix(data=c(1,2,3,4,5,6), nrow=2, ncol=3)
x [,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6x[,-2] [,1] [,2]
[1,] 1 5
[2,] 2 6Liste
Pour accéder aux objets d’une liste on utilisera soit les crochets simples (dans ce cas une liste d’objets est retournée), soit les crochets doubles avec un unique indice (dans ce cas l’objet est extrait) :
seq1<-c(0.2,0.3,0.4,0.1)
L1<-58402
titre<-"phage"
res<-list(titre,L1,seq1)
res[1:2][[1]]
[1] "phage"
[[2]]
[1] 58402res[[3]][1] 0.2 0.3 0.4 0.1Si l’on a donné des étiquettes aux différents objets de la liste (pour obtenir les étiquettes d’une liste, il suffit d’utiliser la fonction ls(res)), alors on peut extraire ces objets en utilisant l’opé- rateur $ :
seq1<-c(0.2,0.3,0.4,0.1)
L1<-58402
titre<-"phage"
res<-list(espece=titre,longueur=L1,composition=seq1)
res$espece[1] "phage"res$longueur[1] 58402res$composition[1] 0.2 0.3 0.4 0.1Tableau de données
Pour fabriquer un sous-tableau de données en sélectionnant certaines colonnes d’un tableau de données, on utilise les crochets simples :
df seq1 seq2 seq3
A 0.2 0.25 0.25
C 0.3 0.35 0.25
G 0.4 0.15 0.40
T 0.1 0.25 0.10df[1:2] seq1 seq2
A 0.2 0.25
C 0.3 0.35
G 0.4 0.15
T 0.1 0.25df[c("seq1","seq3")] seq1 seq3
A 0.2 0.25
C 0.3 0.25
G 0.4 0.40
T 0.1 0.10Si l’on veut seulement récupérer un vecteur colonne du tableau de données, on utilisera la notation matricielle df[,1] ou l’opérateur $ (df$seq1).
Opérateurs et calcul arithmétique
Opérateurs arithmétiques
Les opérateurs arithmétiques agissent aussi bien sur des variables numériques que logiques (dans ce dernier cas, les valeurs logiques sont converties en valeurs numériques : 0 pour FALSE et 1 pour TRUE). Mis à part les opérateurs classiques, addition (+), soustraction (-), multiplication (*) et division (/), on notera les 3 autres opérateurs suivants :
^: puissance%%: modulo%/%: division entière
Ces opérateurs peuvent aussi s’appliquer à des vecteurs (et à des matrices) ; dans ce cas, attention, l’opération est effectuée composante par composante :
x<-1:10
x^2 [1] 1 4 9 16 25 36 49 64 81 100y<-1:10
x+y [1] 2 4 6 8 10 12 14 16 18 20x*y [1] 1 4 9 16 25 36 49 64 81 100Si les deux vecteurs n’ont pas la même taille le plus petit sera “recyclé” autant de fois que nécessaire (avec un warning message)
x<-1:10
y<-1:3
x+y [1] 2 4 6 5 7 9 8 10 12 11Opérateurs de comparaison
Les opérateurs de comparaison s’appliquent à deux objets de n’importe quel mode et re- tournent une valeur logique. En fait les opérateurs opèrent sur chaque élément des objets com- parés et retourne donc un objet logique de même taille :
x<-1:10
y<- (-3:6)^2
x [1] 1 2 3 4 5 6 7 8 9 10y [1] 9 4 1 0 1 4 9 16 25 36x<=y [1] TRUE TRUE FALSE FALSE FALSE FALSE TRUE TRUE TRUE TRUEL’opérateur == (resp !=) vérifie l’égalité (resp. la différence) de deux objets, élément par élément ; il retourne donc un vecteur logique :
x<-seq(2,10,by=2)
y<- 2 * (1:5)
x==y[1] TRUE TRUE TRUE TRUE TRUEPour effectuer une comparaison globale d’objets, la fonction équivalente est identical(x,y) :
x<-seq(2,10,by=2)
y<- 2 * (1:5)
identical(x,y)[1] TRUECette fonction (ainsi que l’opérateur ==) compare la représentation interne des données et retourne TRUE si les objets sont strictement identiques. On se méfiera ainsi des erreurs numériques dues aux calculs numériques sur ordinateur :
(1.1-0.2)==0.9[1] FALSEPour y remédier, la fonction all.equal compare l’égalité approximative, c’est-à-dire avec un seuil de tolérance (qui par défaut dépend des paramètres de la machine), entre deux objets :
all.equal(1.1-0.2,0.9)[1] TRUEOpérateurs logiques
Les opérateurs logiques s’appliquent à un ou deux objets de mode logique et retournent une ou plusieurs valeurs logiques :
!x: NON logiquex&y: ET logiquex|y: OU logiquexor(x,y): OU exclusif
Fonctions mathématiques simples
Il existe un nombre très important de fonctions pour manipuler des données numériques (généralement sous la forme de vecteurs). Outre les fonctions mathématiques de base du type log, exp, cos, abs, sqrt etc. en voici quelques-unes assez courantes :
sum(x),prod(x): somme, produit des éléments dex,min(x),max(x): minimum, maximum des éléments dex,which.min(x),which.max(x): indice du min, max des éléments dex(seule la première occurrence est considérée),round(x,digits=2): arrondi dexavec un nombre de chiffres après la virgule défini par l’option digits,floor(x),ceiling(x): parties entières inférieures ou supérieures dex,length(x): nombre d’éléments dex,rev(x): inverse l’ordre des éléments dex,sort(x,decreasing=F): trie les éléments dexdans l’ordre croissant,choose(n,k): combinaisons de k éléments parmi n.
Par exemple :
x<-choose(6,0:6)
x[1] 1 6 15 20 15 6 1sum(x)[1] 64max(x)[1] 20which.max(x)[1] 4sort(x,decreasing=TRUE)[1] 20 15 15 6 6 1 1En voici d’autres à connotation statistique :
mean(x): moyenne des éléments dex,median(x): médiane des éléments dex,var(x): variance des éléments dex,cov(x,y): covariance entrexety,cor(x,y): corrélation entrexety,sample(x,size=n,replace=FALSE): tirage aléatoire sans remise de n éléments parmi ceux dex.
Calcul matriciel
R offre des facilités pour le calcul matriciel. En voici quelques unes :
x%*%y: produit de deux matrices,t(x): transposée dex,diag(x): extrait la diagonale d’une matricex(dans ce cas renvoie un vecteur) ou crée une matrice diagonale de diagonalex,solve(x): matrice inverse dex,solve(A,b): résout le système linéaire Ax = b,eigen(x): calcule les valeurs et vecteurs propres dex.
Entrées et sorties
Pour les lectures et écritures de données dans des fichiers, R utilise le répertoire de travail. Il est donc nécessaire de préciser le chemin d’accès au fichier si celui-ci n’est pas dans le répertoire de travail.
Liste des données dans un fichier
Nous nous intéresserons ici à la lecture de fichiers ASCII. Les deux principales fonctions sont read.table et scan.
read.table
Cette fonction crée un tableau de données directement à partir des données disposées dans un fichier sous la forme d’un tableau. Par exemple, prenons le fichier virus-count-tri.tab présent dans le répertoire data. Celui-ci est composé de 67 lignes et 87 colonnes : la première ligne est une ligne de commentaire (qu’il conviendra d’ignorer au moment de la lecture les don- nées grâce à l’option skip=1), la deuxième ligne contient la légende des 86 colonnes (virus), la 3ème ligne donne la longueur des séquences de virus et les 64 lignes suivantes correspondent aux 64 trinucléotides. La première colonne du tableau contient les légendes de lignes. La commande suivante :
mydata<-read.table(file="initiation_r_data/virus-count-tri.tab",header=TRUE, skip=1)crée donc un tableau de données nommé mydata de dimension 65 × 86 :
dim(mydata)[1] 65 86dont les colonnes sont nommées selon la première ligne du fichier (header=TRUE) et les lignes selon la première colonne du fichier. Ces noms peuvent s’obtenir en utilisant les fonctions colnames et rownames :
colnames(mydata) [1] "virus01.sq" "virus02.sq" "virus03.sq" "virus04.sq" "virus05.sq"
[6] "virus06.sq" "virus07.sq" "virus08.sq" "virus09.sq" "virus10.sq"
[11] "virus11.sq" "virus12.sq" "virus13.sq" "virus14.sq" "virus15.sq"
[16] "virus16.sq" "virus17.sq" "virus18.sq" "virus19.sq" "virus20.sq"
[21] "virus21.sq" "virus22.sq" "virus23.sq" "virus24.sq" "virus25.sq"
[26] "virus26.sq" "virus27.sq" "virus28.sq" "virus29.sq" "virus30.sq"
[31] "virus31.sq" "virus32.sq" "virus33.sq" "virus34.sq" "virus35.sq"
[36] "virus36.sq" "virus37.sq" "virus38.sq" "virus39.sq" "virus40.sq"
[41] "virus41.sq" "virus42.sq" "virus43.sq" "virus44.sq" "virus45.sq"
[46] "virus46.sq" "virus47.sq" "virus48.sq" "virus49.sq" "virus50.sq"
[51] "virus51.sq" "virus52.sq" "virus53.sq" "virus54.sq" "virus55.sq"
[56] "virus56.sq" "virus57.sq" "virus58.sq" "virus59.sq" "virus60.sq"
[61] "virus61.sq" "virus62.sq" "virus63.sq" "virus64.sq" "virus65.sq"
[66] "virus66.sq" "virus67.sq" "virus68.sq" "virus69.sq" "virus70.sq"
[71] "virus71.sq" "virus72.sq" "virus73.sq" "virus74.sq" "virus75.sq"
[76] "virus76.sq" "virus77.sq" "virus78.sq" "virus79.sq" "virus80.sq"
[81] "virus81.sq" "virus82.sq" "virus83.sq" "virus84.sq" "virus85.sq"
[86] "virus86.sq"rownames(mydata) [1] "long" "aaa" "aac" "aag" "aat" "aca" "acc" "acg" "act" "aga"
[11] "agc" "agg" "agt" "ata" "atc" "atg" "att" "caa" "cac" "cag"
[21] "cat" "cca" "ccc" "ccg" "cct" "cga" "cgc" "cgg" "cgt" "cta"
[31] "ctc" "ctg" "ctt" "gaa" "gac" "gag" "gat" "gca" "gcc" "gcg"
[41] "gct" "gga" "ggc" "ggg" "ggt" "gta" "gtc" "gtg" "gtt" "taa"
[51] "tac" "tag" "tat" "tca" "tcc" "tcg" "tct" "tga" "tgc" "tgg"
[61] "tgt" "tta" "ttc" "ttg" "ttt" Comme on l’a vu précédemment, on accède à la colonne virus05.sq du tableau de données mydata en tapant :
mydata$virus05.sq [1] 39630 1213 743 757 1175 666 410 482 635 798 374 409
[13] 795 1129 624 639 1276 799 434 423 738 527 300 159
[25] 467 366 162 166 537 584 530 399 867 876 358 515
[37] 644 452 183 177 312 522 169 244 320 818 427 411
[49] 728 1000 658 682 1111 749 560 414 966 706 418 436
[61] 732 920 1108 843 1574Le résultat est un vecteur, tout comme mydata[,5].
Par défaut, header=FALSE, ce qui implique que chaque colonne est nommée par défaut V1, V2, etc. Si le fichier ne contient pas les labels de lignes et/ou colonnes, on peut les spécifier au moment de la lecture du tableau de données en utilisant les options row.names/col.names de la fonction read.table. On peut aussi les modifier après coup en utilisant les fonctions rownames() et colnames(), par exemple :
colnames(mydata)<-paste(sep="", "virus",1:86)Si les lignes du fichier de données n’ont pas toutes le même nombre de valeurs, un message d’erreur apparaîtra aussitôt après la commanderead.table. Par défaut, le symbole séparant les décimales est le point, mais on peut le modifier via l’option dec ; par exemple, on pourra spécifier dec="," si besoin. De la même façon, le symbole par défaut permettant de séparer les colonnes est l’espace ; si ce n’est pas le cas, on peut le spécifier via l’option sep, par exemple, sep="|".
scan
Une autre possibilité pour lire un fichier de données est d’utiliser la fonction scan. La principale différence est que scan crée un vecteur numérique par défaut (et non un tableau de données), ou une liste de vecteurs si on lui spécifie leurs modes via l’option what. Cela suppose que chaque colonne contienne des valeurs de même mode. Par exemple, supposons que le fichier short-virus.tab (répertoire initiation_r_data) ne contienne que les 10 premières lignes du fichier initiation_r_data/virus-count-tri.tab (la longueur et les comptages des 9 premiers trinucléotides) pour seulement les 3 premiers virus, sans les labels de lignes (on verra plus loin comment a été crée ce fichier). Alors en tapant
myshortdata<-scan("initiation_r_data/short-virus.tab",what=list(0,0,0),skip=1)
myshortdata[[1]]
[1] 23814 728 453 588 732 431 234 141 485 571
[[2]]
[1] 20869 760 422 553 503 544 223 145 396 726
[[3]]
[1] 31787 833 531 463 1087 695 294 310 836 390on obtient une liste (myshortdata) formée de 3 vecteurs en mode numérique (d’où les 0 dans l’option what ; mettre “” si le vecteur est en mode caractère). L’option skip=1 permet de spécifier le nombre de lignes du fichier à sauter avant d’enregistrer les données (en général des commentaires mais aussi, comme ici, les labels en mode caractère des colonnes). On peut spécifier les noms (ou étiquettes) de ces 3 objets :
myshortdata<-scan("initiation_r_data/short-virus.tab",what=list(seq1=0,seq2=0,seq3=0),skip=1)
myshortdata$seq1
[1] 23814 728 453 588 732 431 234 141 485 571
$seq2
[1] 20869 760 422 553 503 544 223 145 396 726
$seq3
[1] 31787 833 531 463 1087 695 294 310 836 390En pratique, si le fichier contient une unique colonne, on privilégiera scan à read.table en spécifiant what=character() ou what=logical() si besoin.
Afficher des résultats
Affichage à l’écran
On a vu que la façon la plus simple d’afficher le contenu d’un objet à l’écran était de taper son nom suivi de la touche “Entrée”. C’est équivalent à écrire explicitement print(x) :
x<-choose(6,0:6)
print(x)[1] 1 6 15 20 15 6 1On utilisera print(x) à l’intérieur d’une boucle, d’une fonction ou d’un programme. Si l’on veut un “bel” affichage, on peut alors utiliser la fonction cat qui va convertir le contenu des objets en chaî̂ne de caractères (donc sous la forme d’un vecteur), concaténer autant de chaînes de caractères que demandées et imprimer la chaîne résultante :
x<-choose(6,0:6)
cat("Les combinaisons parmi 6 elements sont : ",x,"\n")Les combinaisons parmi 6 elements sont : 1 6 15 20 15 6 1 La chaîne de caractères "\n" signifie un retour à la ligne. Par défaut, la concaténation des chaînes de caractères se fait avec le séparateur sep=" ". La fonction cat est donc particulièrement adaptée à l’affichage de vecteurs (éventuellement de longueur 1). Pour afficher un tableau (matrice ou tableau de données) on utilisera print.
Affichage dans un fichier
Si l’on veut imprimer des résultats dans un fichier, on peut procéder de deux manières :
- Soit rediriger la sortie standard (c’est-à-dire la console) dans un fichier via la fonction
sink(file="file.res", append=TRUE)avant d’afficher ; si l’optionappendn’est pas spécifiée (FALSEpar défaut), l’écriture écrase l’ancien contenu du fichier. Ne pas oublier de rebasculer la sortie standard après écriture en faisantsink().
sink("toto")
cat("Combinaisons parmi 6 elements : \n")
print(x)
sink()Le fichier toto a été créé et contient :
Combinaisons parmi 6 elements :
[1] 1 6 15 20 15 6 1- Soit en utilisant l’option
file(etappend) de la fonctioncat.
cat(file="toto","x=",x,append=TRUE)Le fichier toto contient maintenant :
Combinaisons parmi 6 elements :
[1] 1 6 15 20 15 6 1
x= 1 6 15 20 15 6 1Si l’on veut plutôt écrire le contenu d’un objet, plus précisément un tableau de données ou une matrice, dans un fichier (pour former un nouveau fichier de données par exemple), on pourra utiliser la fonction write.table. Celle-ci stocke l’objet sous la forme d’un tableau de données et le fichier créé pourra alors être relu avec read.table. Par exemple, le fichier short-virus.tab qui a été utilisé précédemment a été obtenu de la façon suivante à partir du tableau de données mydata :
write.table(mydata[1:10,1:3],file="short-virus.tab",row.names=FALSE,quote=FALSE)L’option row.names indique si le nom des lignes doit être écrit dans le fichier (col.names pour les colonnes), tandis que l’option quote indique si les chaînes de caractères sont écrites avec leurs guillemets.
Sauvegarder des données
La sauvegarde des données n’est pas automatique, toutefois, on a la possibilité de sauvegarder l’ensemble des objets en mémoire avant de quitter une session R. Pour cela, il suffit de répondre ’y’ à la question posée suite à la commande q() : ”Save workspace image ? [y/n/c]”. Les objets sont alors sauvegardés dans le répertoire .RData du répertoire de travail et seront disponibles lors de la prochaine session R. On peut néanmoins sauvegarder explicitement des objets R dans un fichier en utilisant la fonction save(mydata,file="mydata.Rdata") (à noter : le fichier mydata.Rdata n’est pas lisible à l’oeil). Pour recharger l’objet enregistré lors d’une nouvelle session, il suffira de faire load("mydata.Rdata").
Programmation
Boucles
Boucle for
La boucle for est utilisée pour répéter une suite d’instructions un nombre prédéfini de fois. La syntaxe standard est la suivante :
for (compteur in suite) {
instructions
}Les accolades ne sont pas nécessaires s’il n’y a qu’une instruction. Voici un exemple ; pour calculer la quantité \(\sum_{i = 1}^{10} i^2\), on effectuera
somme<-0
for (i in 1:10){
somme <- somme + i^2
}
somme[1] 385Boucle while
La boucle while est utilisée pour répéter une suite d’instructions tant qu’une condition est vraie. La syntaxe standard est la suivante :
while (condition) {
instructions
}Voici un exemple ; on souhaite trouver le plus grand entier dont le carré est strictement plus petit que 999 :
entier<-0
while ( (entier+1)^2 < 999 ){
entier <- entier + 1
}
entier[1] 31Tests
Un test si alors permet d’exécuter des instructions seulement si une condition est réalisée, tandis qu’un test si alors sinon permet d’exécuter des instructions différentes selon qu’une condition est vraie ou fausse. La syntaxe standard est la suivante :
if (condition) {
instructions
}else{
instructions
}Voici un exemple :
x<-rpois(1,20)
if (x%%2==0){
pair<-TRUE
}else{
pair<-FALSE
}
x[1] 21pair[1] FALSEL’instruction sinon n’est pas obligatoire mais si elle existe alors le mot-clé else doit être sur la même ligne que l’accolade fermante du si. On peut imbriquer plusieurs boucles si alors sinon si alors sinon etc.
Vectorisation
Comme on l’a déjà vu, un gros avantage de R est sa capacité à travailler vectoriellement ce qui évite d’avoir à programmer des boucles for et/ou des tests. Ainsi, la boucle for vue précédemment peut être remplacée simplement par
x<-1:10
y<-x^2tandis que la boucle suivante :
n<-10
x<-rpois(n,20)
pair<-rep(FALSE,n)
for (i in 1:n){
if (x[i]%%2==0) pair[i]<-TRUE
}peut s’écrire simplement
pair[x%%2==0]<-TRUEPlus généralement, toute fonction (de R ou créée par l’utilisateur) peut être appliquée à tous les éléments d’une matrice (ou seulement à certaines lignes ou colonnes), d’un vecteur ou d’une liste. Pour cela on utilise les fonctions apply, sapply ou lapply.
Écrire une fonction
L’essentiel du travail sous R se fait à l’aide de fonctions dont les arguments sont indiqués entre parenthèses. Outre les fonctions déjà implémentées dans R (on en a déjà vu un certain nombre), l’utilisateur peut écrire ses propres fonctions (c’est même d’ailleurs conseillé !) afin de pouvoir efficacement répéter certaines opérations. La syntaxe standard est la suivante :
function name<-function(arg1, arg2,. . ., argn) {
instructions
return(result)
}Voici par exemple l’écriture de la fonction zscore qui prend 3 arguments, une observation obs (éventuellement plusieurs sous la forme d’un vecteur), une moyenne exp (éventuellement un vecteur) et une variance var (éventuellement un vecteur), et retourne le z-score associé c’est-à-dire \((obs - exp)\sqrt{var}\) :
zscore<-function(obs,exp,var)
{
res<- (obs-exp)/sqrt(var)
return(res)
}Pour pouvoir exécuter cette fonction, il faut la charger en mémoire, soit en tapant chacune des lignes au clavier soit en l’écrivant dans un fichier et en “compilant” ce dernier avec la fonction source, comme pour un programme (paragraphe suivant). Dès que la fonction est chargée, on l’utilise naturellement :
x<-c(3,5)
xmoyenne<-c(2.5,4.8)
xvariance<-c(0.7,0.9)
score<-zscore(obs=x,exp=xmoyenne,var=xvariance)
score[1] 0.5976143 0.2108185Dans cet appel à la fonction zscore, le nom des arguments (obs, exp et var) a été utilisé (fortement recommandé) ce qui permet par exemple de passer leur valeur dans le désordre. On peut ommettre le nom des arguments, en appelant alors zscore(x,xmoyenne,xvariance), mais dans ce cas il faut absolument respecter l’ordre des arguments.
Les variables définies dans les fonctions (res dans la fonction zscore par exemple) sont considérées comme des variables locales. Si ces variables existaient déjà dans l’environnement “extérieur” à la fonction alors leur contenu n’est pas modifié. Si elles n’existaient pas, alors elles n’existeront pas plus après l’appel à la fonction. Par contre, si la fonction utilise une variable non définie dans la fonction, alors R va la chercher dans l’environnement extérieur ; si elle existe, R utilise sa valeur, sinon un message d’erreur est donné.
Le code des fonctions créées et chargées en mémoire s’obtient simplement en tapant le nom de la fonction suivi de “entrée”.
Écrire un programme
On écrira typiquement un programme R dans un fichier texte ayant l’extension ’.r’, par exemple myprog.r. Si le programme définit des fonctions et les utilise, il convient de les définir avant de les utiliser (par exemple en tête du fichier, éventuellement après l’assignation des variables globales). Le caractère ’#’ sert à ajouter des commentaires : en effet, R passe à la ligne suivante. Pour exécuter le programme contenu dans le fichier myprog.r, il suffit de taper
source("myprog.r")Graphiques
R offre de nombreuses possibilités pour effectuer des graphiques et les multiples options graphiques permettent une grande flexibilité. La commande demo(graphics) donne un aperçu de ces possibilités. Ici nous allons nous contenter de voir les représentations graphiques les plus classiques (graphe 2D, histogramme, boxplot) ainsi que les options graphiques les plus courantes. Pour chaque fonction, la liste complète des options s’obtient via l’aide-en-ligne.
Généralement, l’affichage d’un graphique se fait dans une fenêtre graphique que l’on ouvre avec la commande
x11()La fermeture de la fenêtre graphique se fait avec la commande :
dev.off()On verra plus loin que le graphique peut aussi être sauvegardé ou directement effectué dans un périphérique graphique de type fichier.
Graphe 2D
Nuage de points
Nous allons nous mettre ici dans le cas où nous avons un certain nombre de points à représenter en dimension 2, c’est-à-dire que chaque point a une abscisse et une ordonnée. En d’autres termes, nous disposons d’un vecteur d’abscisses (x) et un vecteur d’ordonnées associées (y). L’exemple sur lequel nous allons travailler va être le suivant : les points correspondent à des trinucléotides, leur abscisse (resp. ordonnée) est leur comptage dans la première (seconde) séquence du tableau de données mydata :
x<-mydata[-1,1]
y<-mydata[-1,2]
length(x)[1] 64length(y)[1] 64Le graphique de base (cf. Figure 2) s’obtient alors en faisant :
plot(x,y)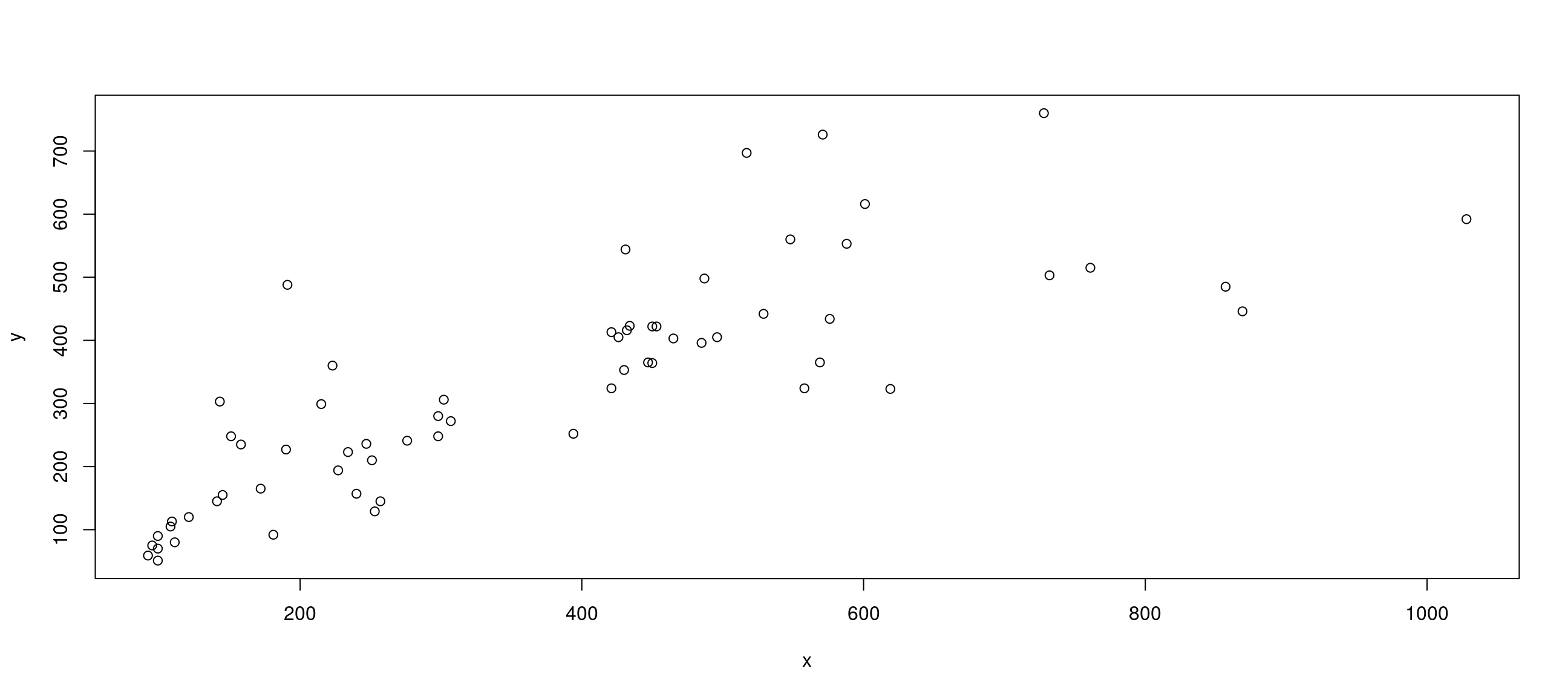
Nous allons modifier ce graphique en jouant sur plusieurs options.
xlim,ylimfixent les limites inférieures et supérieures des axes (vecteurs à deux éléments),xlab,ylabpermettent de spécifier la légende des axes (mode caractère),mainpermet de mettre un titre au dessus du graphique (mode caractère),pchdéfinit le symbole représentant les points : soit un entier de 1 à 25, soit n’importe quel caractère entre guillemets,colspécifie la couleur des symboles ("blue","red"etc. la liste des couleurs est disponible aveccolors()tandis querainbow(n)retourne un vecteur de n couleurs régulièrement réparties dans l’arc-en-ciel) ; on peut aussi jouer sur la couleur du titre (col.main), des labels des axes (col.lab) et de la graduation des axes (col.axis),btycontrôle la forme du cadre : carré par défaut (“o”), en L (“l”), en U (“u”), en C (“c”), en 7 (“7”) ou en crochet (“]”).
Par exemple, la Figure 3 a été obtenue par :
plot(x,y,xlab="Sequence 1",ylab="Sequence 2",main="Comptage des trinucleotides", xlim=c(0,1200),ylim=c(0,1200),pch=18,col="blue",bty="l")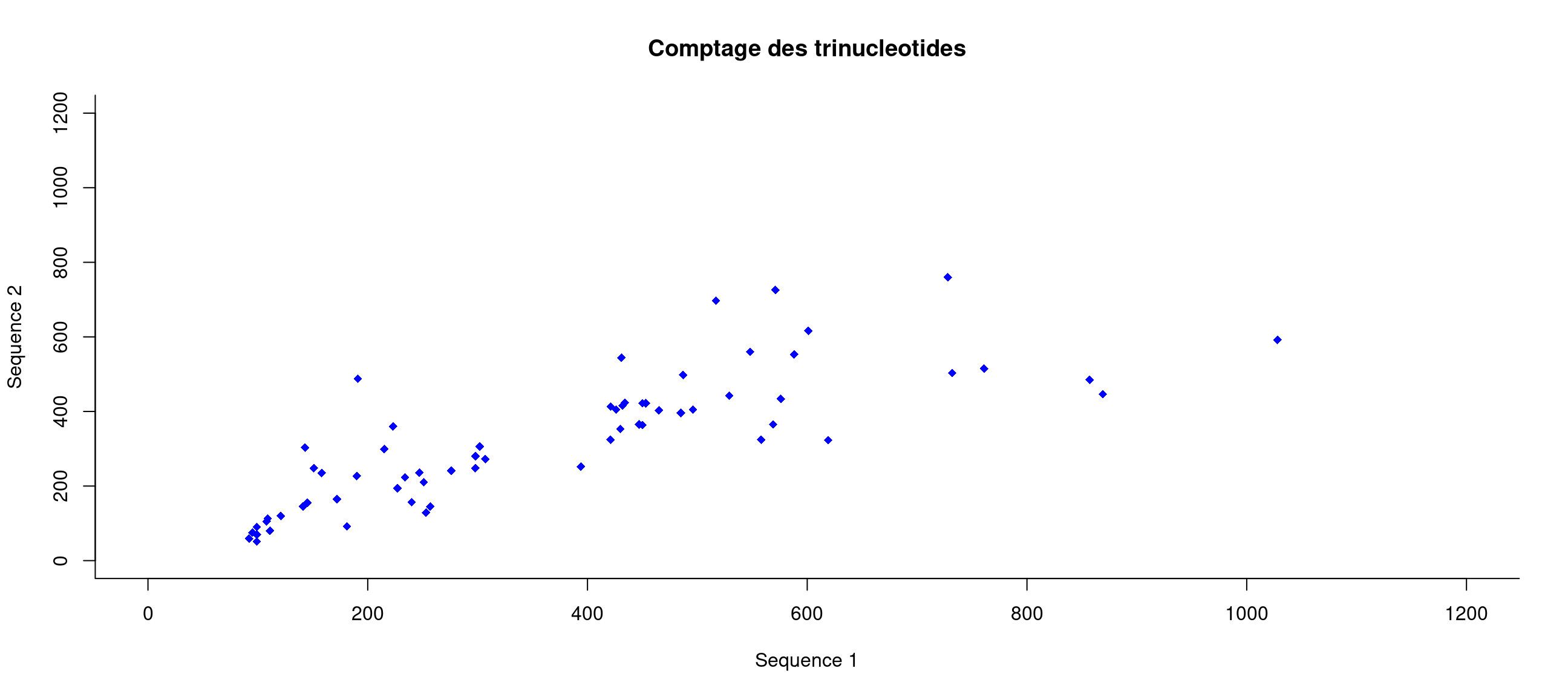
On verra plus loin comment rajouter des éléments graphiques sur cette figure : axes, droites, texte, légende etc.
On peut jouer sur la taille des symboles grâce à l’option cex. Par défaut cex=1 ; on peut lui fournir un nombre positif qui représente un coefficient multiplicatif par rapport à la taille par défaut (une valeur entre 0 et 1 pour réduire la taille, ou plus grande que 1 pour au contraire l’augmenter). De la même façon les options cex.axis, cex.lab et cex.main contrôlent la taille des graduations des axes, des labels des axes et du titre.
Pour changer le style du texte, on utilise l’option font qui se décline aussi sous les formes font.axis, font.lab et font.main, (1 pour normal, 2 pour gras, 3 pour italique et 4 pour gras italique).
Pour utiliser une échelle logarithmique, on utilisera l’option log qui vaudra soit "x", soit "y", soit "xy".
Courbe
Pour dessiner une “courbe”, on utilise aussi la fonction plot à la “seule” différence que l’on souhaite relier les points entre eux. Pour cela l’option principale est type qui vaudra soit "b" si l’on veut des points connectés par une ligne soit "l" si l’on veut seulement une ligne (par défaut type="p").
Comme exemple, nous allons tracer la densité de la loi normale de moyenne 0 et de variance 1 entre -3 et 3 (cf. Figure 4) :
t<-seq(-3,3,by=0.2)
plot(t,dnorm(t,mean=0,sd=1),type="l",col="red",main="densite N(0,1)",font.main=4)
Pour cet exemple, on aurait pu utiliser la fonction curve qui trace la courbe de fonctions de type \(f(x)\) pour lequel R a accès aux valeurs pour tout \(x\). On peut jouer sur le type de ligne à l’aide de l’option lty. Par défaut lty=1 ce qui donne des lignes continues, mais on peut obtenir des tirets (lty=2), des pointillés (lty=3), une alternance de points et tirets (lty=4), des tirets longs (lty=5) ou une alternance de tirets courts et longs (lty=6). On peut aussi jouer sur l’épaisseur des lignes à l’aide de l’option lwd qui vaut 1 par défaut.
Histogramme
Pour tracer un histogramme, la commande de base est la fonction hist (cf. Figure 5) :
hist(x)Voici certaines options spécifiques de la fonction hist :
breaks: permet de spécifier les points de ruptures entre les barres de l’histogramme, soit sous la forme d’un vecteur, soit sous la forme d’un nombre de barres.freq: permet de choisir l’histogramme des effectifs, frequency en anglais (freq=TRUE, option par défaut), ou celui des proportions (freq=FALSE).col: indique la couleur pour remplir les barres.plot: siplot=FALSE, l’histogramme n’est pas tracé et la fonction renvoie la liste des points de ruptures et les effectifs.right: permet de choisir des intervalles de la forme ]a, b] siright=TRUE(par défaut ils sont de la forme [a, b[).
par(mfrow=c(1,2))
hist(x)
hist(x,breaks=15,freq=FALSE,col="blue",main="Histogramme des comptages des trinucleotides")
par(mfrow=c(1,1))
Boxplot
La représentation en “boîte à moustache” d’un vecteur est à mi-chemin entre un histogramme et la fonction de répartition. Elle permet de visualiser la moyenne, la médiane, les quartiles à 25% et 75% de la distribution empirique. Elle s’obtient avec la fonction boxplot. Voici comment obtenir le boxplot de nos deux vecteurs x et y (cf. Figure 6) :
boxplot(list(x,y))
Par défaut, les moustaches ont une longueur maximale égale à 1.5 fois la taille de la boite. Ce coefficient peut être modifié avec l’option numérique range. On peut aussi modifier la largeur de boîte avec l’option width (c’est un vecteur aussi long qu’il y a de boxplots à afficher). L’option names de type caractère permet de spécifier les labels à afficher sous chaque boîte, par exemple ici on pourrait utiliser names=c("x","y")).
Fonctions graphiques secondaires
Voici d’autres fonctions graphiques dites secondaires car elles agissent sur un graphique existant.
abline(a,b): trace la droite \(y = bx+a\). Pour tracer une droite horizontale (resp. verticale), on pourra utiliserabline(h=)(resp.abline(v=)).segments(x,y,x’,y’): trace le segment entre le point de coordonnées (\[x, y\]) et le point de coordonnées (\(x’, y’\)).points(x,y,...): ajoute des points,lines(x,y,...): relie de nouveaux points,text(x,y,labels,...): ajoute le texte défini par labels au point de coordonnées (\(x,y\)).mtext(text,side=3,line=0,...): ajoute le texte défini par text dans la marge (du bas si side=1, de gauche si side=2, du haut si side=3, de droite si side=4) et à un nombre de lignes du cadre spécifié par line.legend(x,y,legend,...): ajoute une légende au point de coordonnées (\(x,y\)).
Cette liste n’est pas exhaustive.
Voici un premier exemple (cf. Figure 7) qui reprend la Figure 4 :
trinucleotides<-rownames(mydata)[-1]
plot(x,y,xlab="Sequence 1",ylab="Sequence 2",main="Comptage des trinucleotides", xlim=c(0,1200),ylim=c(0,1200),type="n")
text(x,y,labels=trinucleotides,col="green")
abline(h=0)
abline(v=0)
abline(a=0,b=1)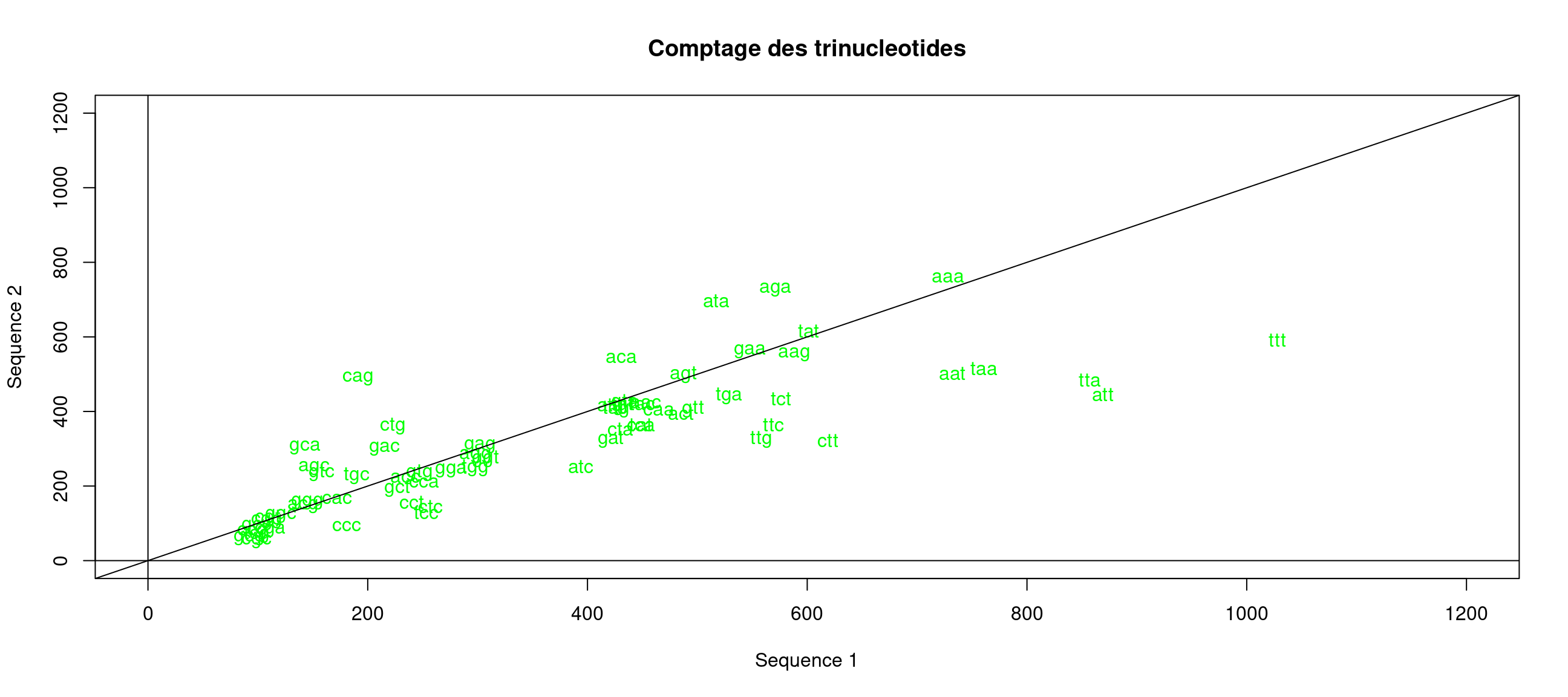
Notez l’option type="n" qui affiche les points de façon invisible.
Voici un deuxième exemple (cf. Figure 8) qui reprend la Figure 5 :
t<-seq(-3,3,by=0.2)
plot(t,dnorm(t,mean=0,sd=1),ylab="densite", type="l",col="red")
points(t,dnorm(t,mean=0,sd=sqrt(2)),type="l",lty=2,col="blue")
legend(1.5,0.35,legend=c("N(0,1)","N(0,2)"),lty=c(1,2),col=c("red","blue"))
mtext(side=3,line=1,"Densites gaussiennes")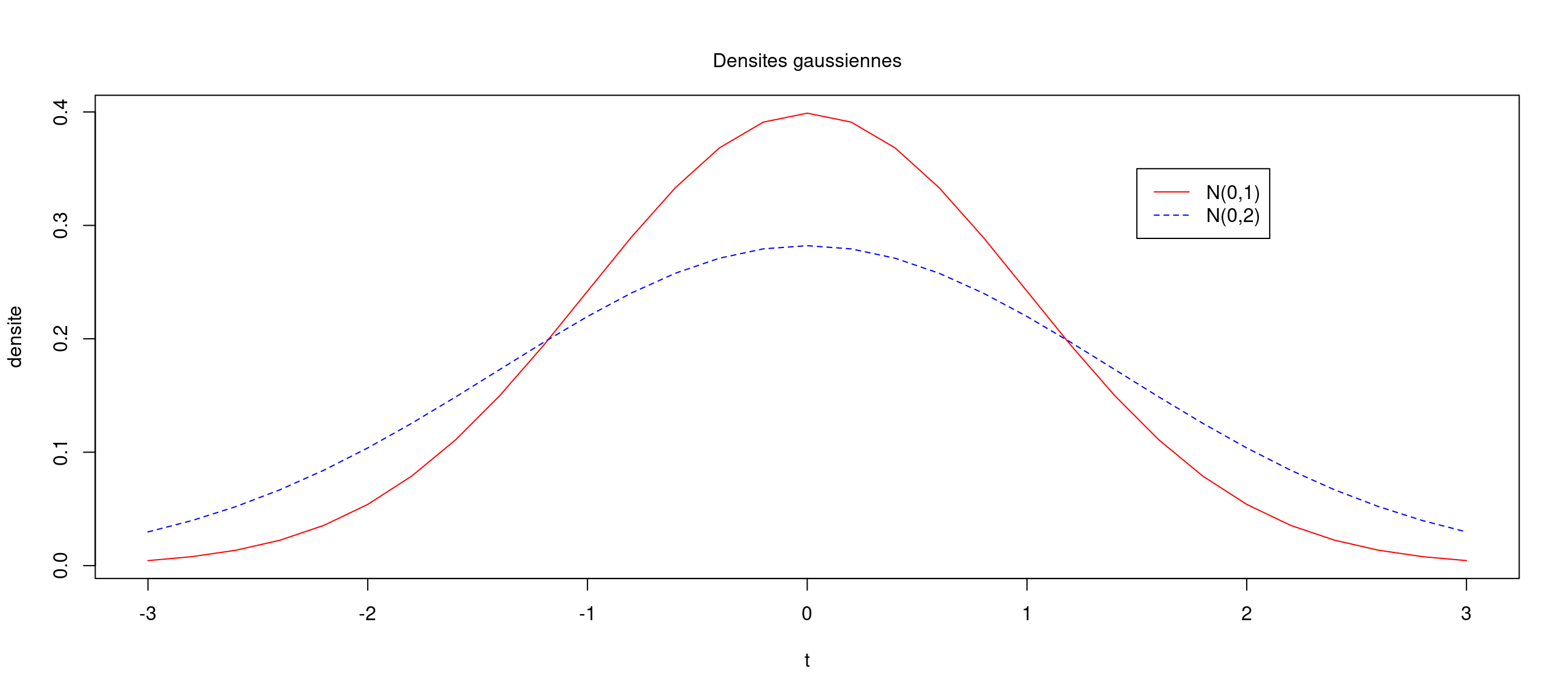
Paramètres graphiques
Certaines options des fonctions graphiques vues précédemment (cex, col, font, lty, lwd, pch, bty etc.) sont en fait des paramètres graphiques qui peuvent être modifiés indépendamment de ces fonctions. Néanmoins, il existe d’autres paramètres graphiques qui ne peuvent pas être utilisés comme options de fonctions graphiques. Le nombre de paramètres graphiques est élevé (leur liste s’obtient en tapant ?par).
Nous en citerons 3 nouveaux :
bg: spécifie la couleur de l’arrière-plan,mfcol: un vecteur de la forme c(nrow,ncol) qui partitionne la fenêtre graphique en nrow lignes et ncol colonnes ; les graphiques s’affichent alors successivement par colonne,mfrow: analogue demfcolavec un affichage par ligne.
De manière générale, les paramètres graphiques se modifient via la fonction par. Par exemple, la commande
par(bg="lightsalmon")a pour effet d’attribuer un fond saumon aux prochains graphiques.
Avant de modifier les paramètres graphiques, il est important de sauvegarder les valeurs initiales de façon à pouvoir les rétablir par la suite.
Périphériques graphiques
Le contenu d’une fenêtre graphique peut être sauvegardé dans un fichier via la fonction dev.copy(device=pdf,file="graphe.pdf") suivie de dev.off().
Les graphiques peuvent aussi directement s’exécuter dans d’autres périphériques que la fenêtre graphique, en particulier dans des fichiers. Ces périphériques sont ouverts avec une fonction qui dépend du format du fichier, par exemple :
postscript(file="graphe.ps")
pdf(file="graphe.pdf")
jpeg(file="graphe.jpg")La liste des périphériques disponibles pour votre installation s’obtient en tapant ?device.
Ces périphériques se ferment de la même façon que pour la fenêtre graphique à savoir avec dev.off().
Le dernier périphérique ouvert devient celui sur lequel les graphiques s’affichent. Si plusieurs périphériques ont été ouverts (et non fermés), leur liste s’obtient par la commande dev.list() et on peut les fermer en spécifiant à dev.off leur numéro.
Quelques analyses statistiques
Nous illustrerons ce chapitre en utilisant deux jeux de données.
Le premier jeu de données provient du tableau symmétrique de celui déjà utilisé au chapitre 2 ; il concerne des comptages de mots (en colonnes) dans des séquences d’ADN de différents virus (en ligne).
mynewdata <- read.table("initiation_r_data/composition3mer.tab",header=T, skip=1)
dim(mynewdata)[1] 86 66colnames(mynewdata) [1] "seq" "long" "aaa" "aac" "aag" "aat" "aca" "acc" "acg" "act"
[11] "aga" "agc" "agg" "agt" "ata" "atc" "atg" "att" "caa" "cac"
[21] "cag" "cat" "cca" "ccc" "ccg" "cct" "cga" "cgc" "cgg" "cgt"
[31] "cta" "ctc" "ctg" "ctt" "gaa" "gac" "gag" "gat" "gca" "gcc"
[41] "gcg" "gct" "gga" "ggc" "ggg" "ggt" "gta" "gtc" "gtg" "gtt"
[51] "taa" "tac" "tag" "tat" "tca" "tcc" "tcg" "tct" "tga" "tgc"
[61] "tgg" "tgt" "tta" "ttc" "ttg" "ttt" Comme nous avons besoin de la fréquence des mots dans la séquence (et non du nombre de mots), nous allons construire, à partir de mynewdata, un nouveau jeu de données Y en divisant le nombre de mots par la longueur de la séquence correspondante.
Y <- cbind(mynewdata[,c(1,2)],mynewdata[,-c(1,2)]/mynewdata[,2])
dim(Y)[1] 86 66Le deuxième jeu de données est extrait d’une expérimentation en nutrition animale ; il concerne l’augmentation de poids de porcelets en fonction du régime alimentaire de leur mère.
porcelets <- read.table("initiation_r_data/poids.txt",header=T)
dim(porcelets)[1] 24 2colnames(porcelets)[1] "aliment" "pds" Statistiques descriptives
Analyse en Composantes Principales (ACP)
L’analyse en composantes principales ou ACP, donne une représentation synthétique, numérique ou graphique, des données. Cette méthode est particulièrement intéressante lorsqu’on dispose d’un nuage de points dans un espace de dimension élevée, c’est-à-dire qu’à chaque observation correspond un grand nombre de variables. L’ACP va conduire à un sous-espace de plus petite dimension, tel que la projection sur ce sous-espace retienne la majeure partie de l’information.
L’ACP s’utilise avec des données quantitatives. Les données sont structurées en lignes, associées aux individus (individu = unité observable), et en colonnes, associées aux variables qui décrivent les individus. Les lignes sont généralement indicées par i, i ∈ {1, …, n} et les colonnes par j, j ∈ {1, …, p}. Si l’on appelle X ce tableau de données, l’élément (i, j) de X, noté x i,j , est égal à la valeur prise par la variable j pour l’individu i. Le principe de l’ACP est d’établir une nouvelle base orthonormée dans laquelle les axes (appelés composantes) sont construits séquentiellement de façon à absorber une quantité maximale de variance. Les nouveaux axes sont ainsi ordonnés, le premier axe portant la plus grande part de la variabilité et le dernier axe portant la plus faible part. L’ACP nécessite une phase de préparation des données qui doivent être centrées. Les variables seront aussi réduites si l’on veut qu’elles aient toutes la même influence dans l’analyse. La fonction scale() permet de centrer et/ou réduire les données en colonnes (à noter : par défaut center et scale valent TRUE) : scale(x, center = TRUE, scale = TRUE). Dans les fonctionnalités chargées par défaut avec R (package stats), 2 fonctions sont disponibles pour réaliser une ACP : princomp() et prcomp().
princomp() calcule les valeurs propres sur la matrice de covariance ou de corrélation alors que prcomp() utilise la décomposition en valeurs singulières ; prcomp est une généralisation de la méthode princomp.
Pour illustrer l’ACP, nous allons sélectionner un sous-ensemble du jeu de données Y :
indd1 <- c(3,4,6,7,9,12,16,21,22,30,31,37,39,44,46,47,53,54,60,66,70,81,82)
mots1 <- c("aaa","agg","cgt","tgt","ttt")
Y1 <- Y[indd1,mots1]
dim(Y1)[1] 23 5Sur ce sous-ensemble, noté Y1, nous allons appliquer une ACP en utilisant la fonction prcomp qui possède les options center et scale fixées respectivement à TRUE et FALSE par défaut. Ici, nous avons choisi de normaliser les données, aussi nous modifions l’option scale :
Y1.prcomp <- prcomp(Y1,scale=T)Nous utilisons ensuite la fonction summary() qui résume les résultats d’une analyse. Cette fonction reconnaı̂t le type d’objet qu’on lui attribue et applique sur l’objet la fonction summary spécifique à cet objet.
summary(Y1.prcomp)Importance of components:
PC1 PC2 PC3 PC4 PC5
Standard deviation 1.6230 1.3900 0.56236 0.30439 0.15789
Proportion of Variance 0.5268 0.3864 0.06325 0.01853 0.00499
Cumulative Proportion 0.5268 0.9132 0.97648 0.99501 1.00000La variabilité totale est représentée par la surface de l’histogramme (Figure 9). Le but de l’analyse étant de conserver le maximum de variabilité avec le minimum de composantes, on observe la décroissance de la variance sur les différentes composantes et on cherche une rupture de la pente. Ainsi, on remarque sur la Figure 9 une rupture de la pente entre la deuxième et la troisième composante. En se rapportant aux valeurs de proportions de variance cumulées, on voit que les deux premières composantes portent 91,3% de la variance. On peut donc choisir de réduire l’espace à ces 2 premières composantes.
La représentation sous forme de biplot (Figure 10) permet de visualiser la proximité entre les individus. Elle permet également d’apprécier les corrélations entre les variables. Par défaut, le nuage est projeté sur les deux premiers axes principaux, mais on peut spécifier explicitement les axes en utilisant l’option choices=c(1,2) (valeur par défaut).
plot(Y1.prcomp)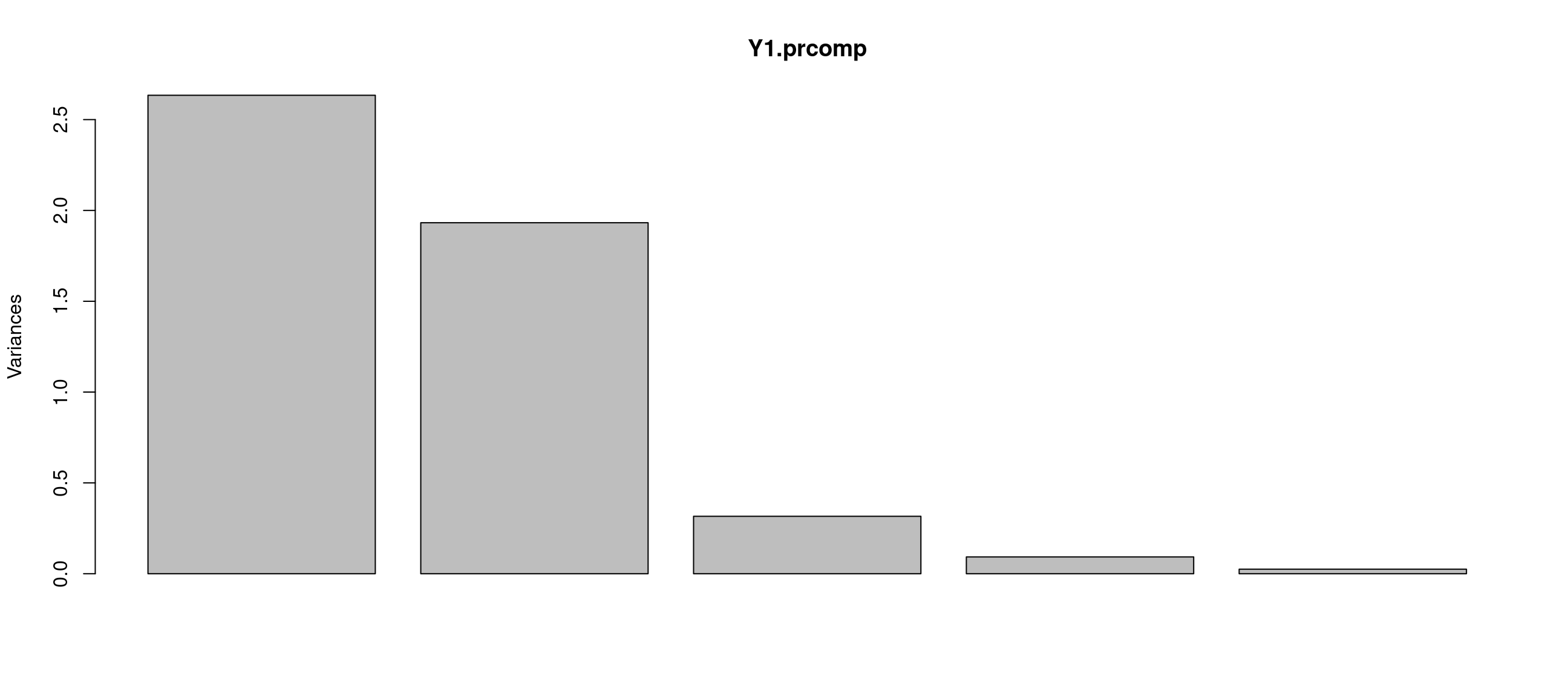
biplot(Y1.prcomp)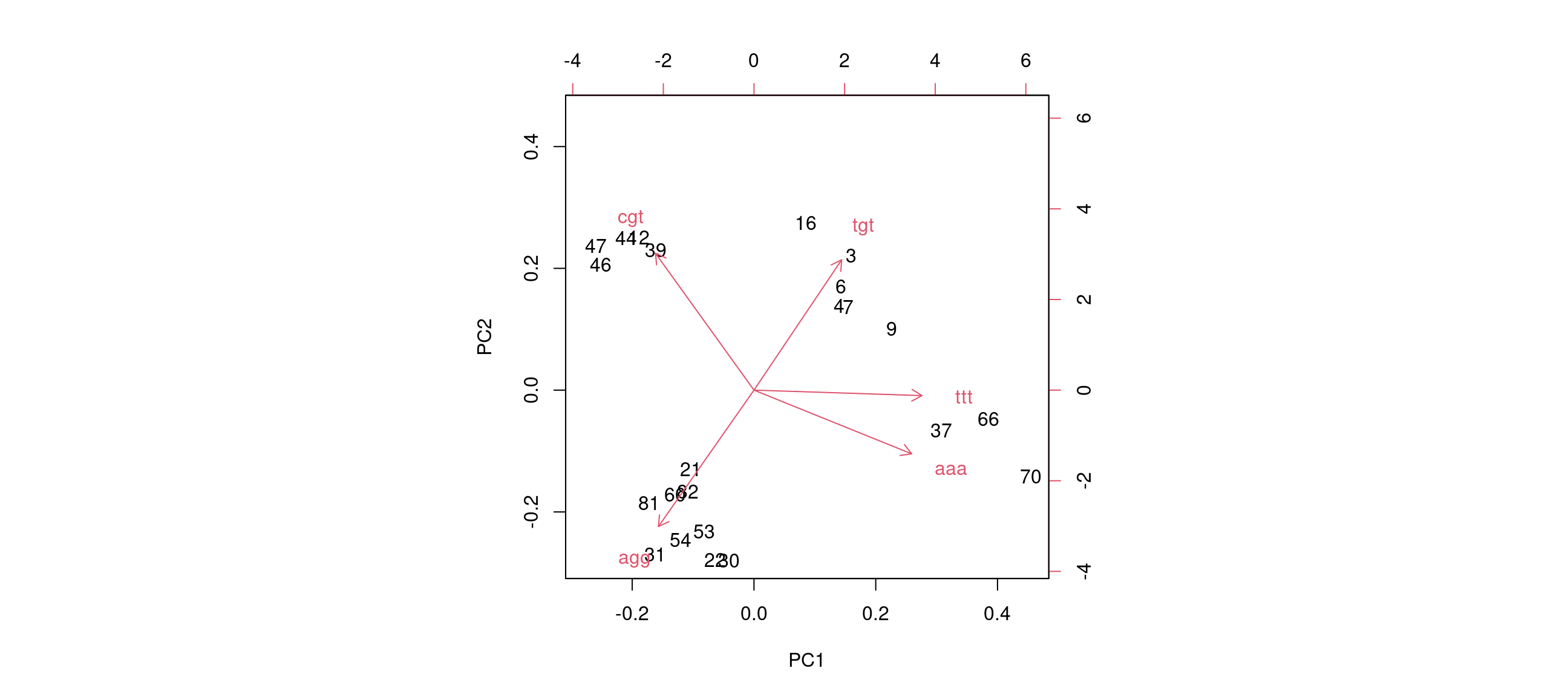
Classification
L’objectif de la classification est de regrouper les objets similaires parmi un ensemble d’objets dont les caractéristiques sont connues. La similarité des objets est appréciée en calculant leurs distances. Plusieurs distances sont disponibles dans R. Il existe aussi plusieurs façons de regrouper les objets : soit par un processus hiérarchique, ascendant ou descendant, soit sur la base du “partitionnement” à partir d’un nombre prédéfini de classes. Dans R, il existe la bibliothèque cluster qui propose plusieurs méthodes de classification adaptées à différents types de données. Par exemple, la fonction agnes() permet de réaliser une classification hiérarchique ascendante : à chaque étape du processus les deux objets ou classes les plus proches sont fusionnés pour former une nouvelle classe, et les distances de cette nouvelle classe à tous les autres, objets ou classes, sont recalculées.
library(cluster)
agnes(Y1)Call: agnes(x = Y1)
Agglomerative coefficient: 0.8894554
Order of objects:
[1] 3 16 4 7 6 9 37 12 44 39 46 47 21 60 82 81 22 31 53 54 30 66 70
Height (summary):
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.001691 0.003807 0.006542 0.012209 0.014176 0.056257
Available components:
[1] "order" "height" "ac" "merge" "diss" "call"
[7] "method" "order.lab" "data" La fonction agnes retourne une liste composée de plusieurs champs, en particulier, le coefficient d’agglomération (ac) qui est une mesure de force de la “structure en classes”. Cette mesure est calculée de la façon suivante : soit d(i) la hauteur de la première fusion de chaque objet et soit h la hauteur de la dernière fusion, le coefficient d’agglomération s’écrit alors comme la moyenne des (1 − d(i)/h). Une valeur proche de 1 du coefficient d’agglomération signifie que les objets sont fortement structurés en classes. Une valeur proche de 0 signifie que les objets appartiennent tous à une même classe.
Le champ “merge” liste les étapes de fusion. Les chiffres affectés du signe “-” correspondent aux objets de départ. Les chiffres sans signes sont les numéros des classes attribués au fur et à mesure qu’elles sont construites.
Le champ “height” est le vecteur des hauteurs de fusion.
Tous les champs peuvent être extraits à l’aide du $. Par exemple :
agnes(Y1)$height [1] 0.007256906 0.011765932 0.001708030 0.003026567 0.022953801 0.014979353
[7] 0.034979462 0.001872430 0.003747635 0.005237884 0.001691141 0.029214443
[13] 0.005472515 0.003984952 0.005827433 0.011098483 0.008916999 0.005466319
[19] 0.003279229 0.018711126 0.056256535 0.011160517Deux types de graphiques sont proposés pour afficher les résultats de la fonction agnes :
- la bannière, qui s’obtient avec la fonction bannerplot() et qui reproduit les hauteurs des différentes fusions dans l’ordre de sortie des objets ;
- l’arbre de classification qui s’obtient avec la fonction
pltree()et qui permet une vue synthétique des regroupements et le repérage d’éventuelles classes.
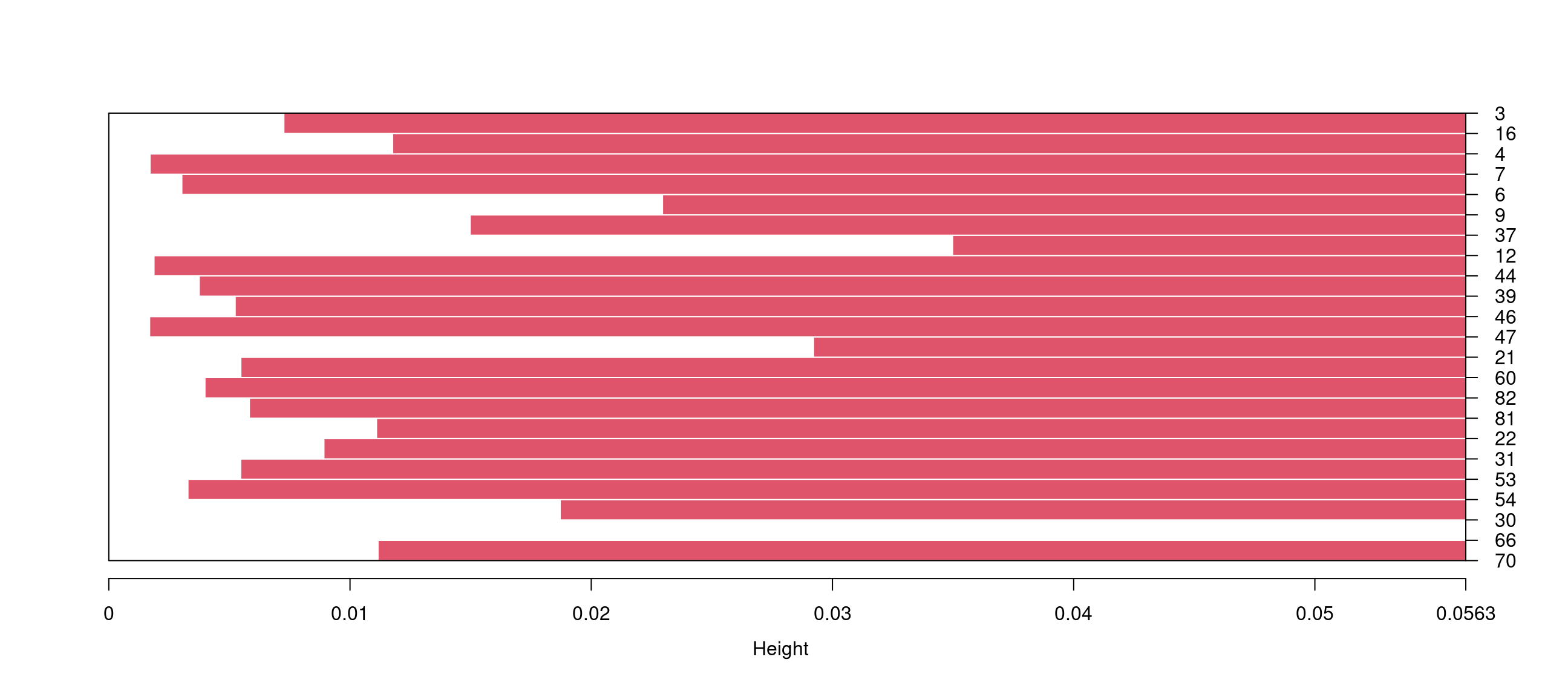
Nous avons représenté sous forme de bannière (Figure 11) puis d’arbre (Figure 12) les résultats de la classification des séquences de Y1.
La classification peut être réalisée à partir des corrélations. Par exemple, les Figure 13 et Figure 14 montrent l’arbre de classification établi pour les séquences de Y1 et celui établi pour les variables de Y, tous deux réalisés à partir des corrélations.
bannerplot(agnes(Y1),main="",sub="")pltree(agnes(Y1))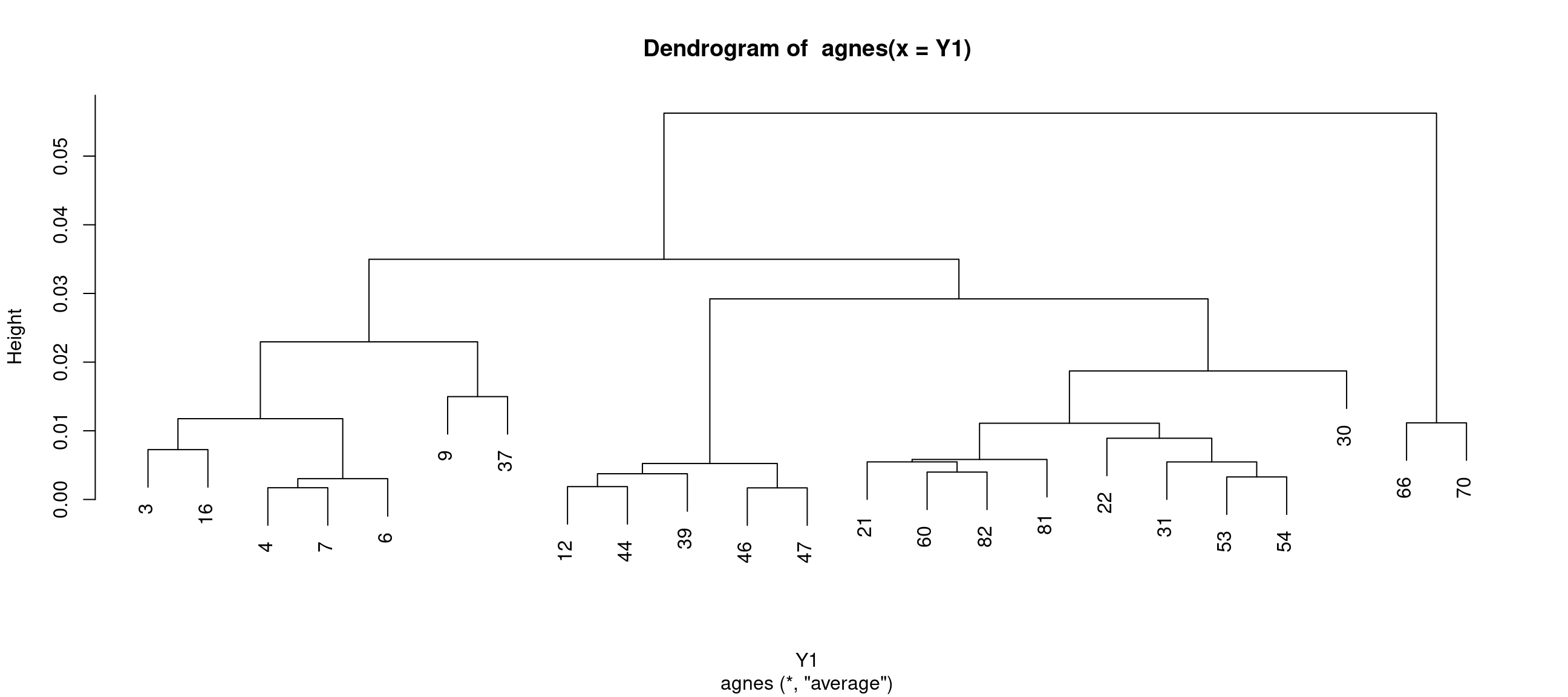
pltree(agnes(cor(t(Y1))))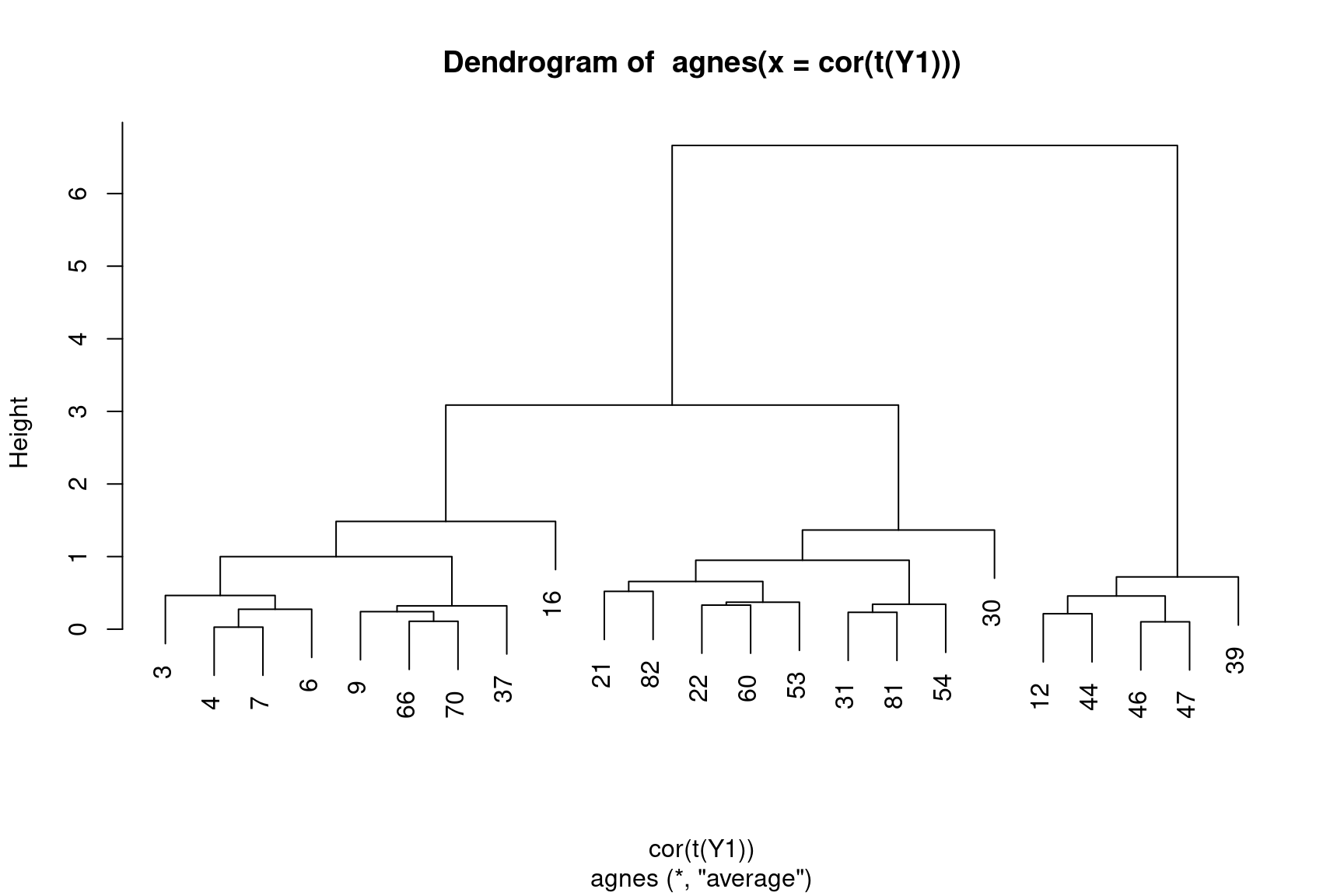
pltree(agnes(cor(Y[,-c(1,2)])))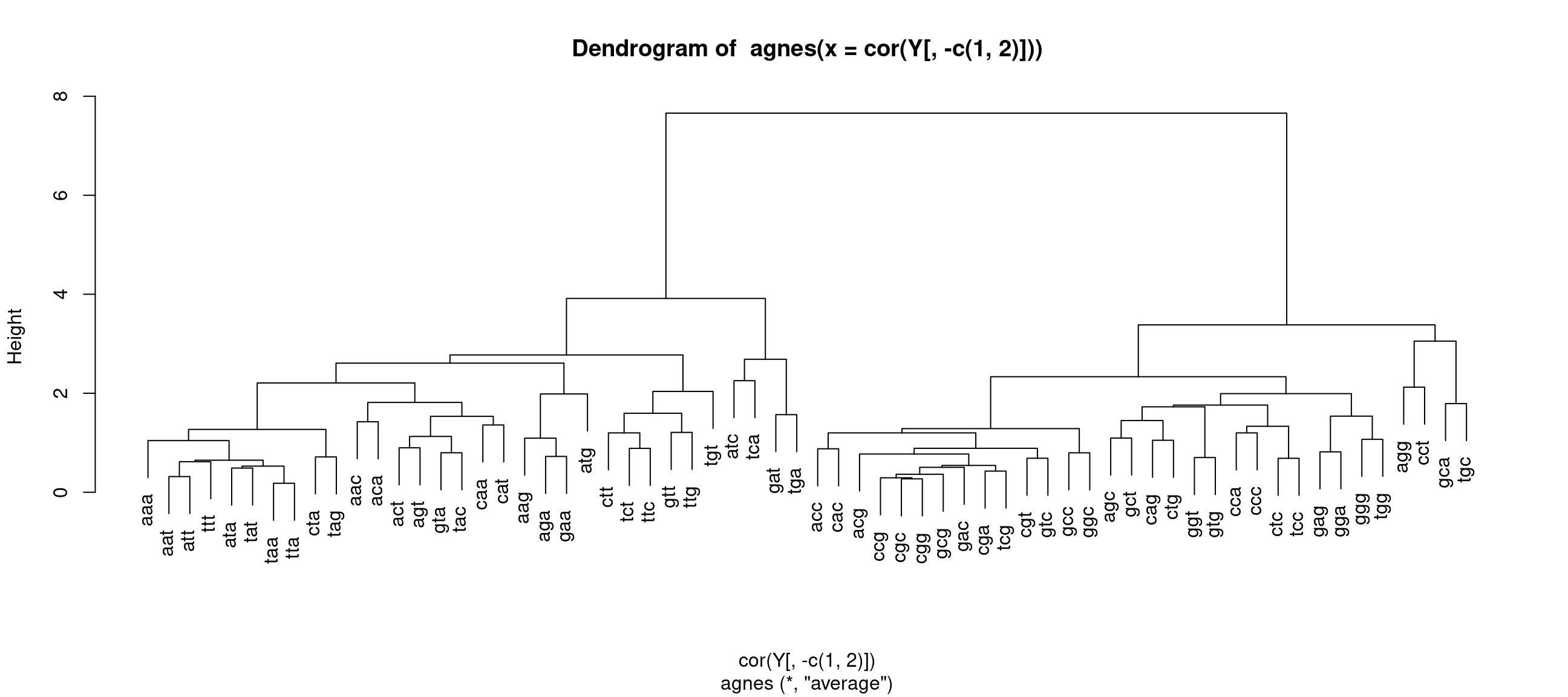
Statistiques inférentielles
La régression linéaire
La régression linéaire est une technique statistique utilisée pour modéliser des relations entre un ensemble de variables prédictives (les prédicteurs) et une ou plusieurs variables “réponse”. Toutes les variables impliquées sont quantitatives. Pour illustrer cette technique, nous considérerons la forme la plus simple avec une seule variable réponse, le mot “aaa”, et un seul prédicteur, le mot “gtg”. Si nous notons y la réponse et x 1 , le prédicteur, le modèle statistique s’écrit de la façon suivante :
\(y = \mu + \alpha x_{1} + \epsilon\)
y = μ + αx 1 + εoù μ représente l’intersection de la droite de régression avec l’axe des ordonnées, α la pente de la droite de régression et ε l’erreur résiduelle.
La fonction R permettant de réaliser une régression linéaire est la fonction lm(). Les arguments principaux attendus par cette fonction sont le modèle, qui en langage R s’écrit : reponse ∼ predicteur, et le nom du jeu de données.
Y.lm <- lm(aaa ~ gtg , data=Y)
summary(Y.lm)
Call:
lm(formula = aaa ~ gtg, data = Y)
Residuals:
Min 1Q Median 3Q Max
-0.022422 -0.007019 -0.002900 0.006263 0.034720
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.062529 0.003566 17.536 < 2e-16 ***
gtg -2.759367 0.280193 -9.848 1.17e-15 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.01074 on 84 degrees of freedom
Multiple R-squared: 0.5359, Adjusted R-squared: 0.5303
F-statistic: 96.98 on 1 and 84 DF, p-value: 1.171e-15Il est habituel de regarder la distribution des résidus afin de vérifier l’hypothèse de normalité sous-jacente au modèle. Cette distribution doit être centrée autour de zéro, et donc la médiane doit être proche de zéro. On peut le vérifier avec le champ Residuals du résumé de l’analyse. Une autre façon de vérifier la normalité est de tracer un Q-Q plot des résidus. Le vecteur des résidus peut être extrait en utilisant soit $resid, soit la fonction resid(). On voit sur la Figure 15 que la linéarité n’est pas parfaite. L’hypothèse de normalité pourrait donc être, dans ce cas, remise en question. Le champ Coefficients fournit l’estimation des valeurs de la pente α et de l’intercept μ ainsi que les statistiques qui leur sont associées. Le vecteur des coefficients peut être extrait en utilisant $coeff. A partir de ces valeurs, on peut tracer la droite estimée sur le graphique représentant aaa en fonction de gtg (Figure 15).
qqnorm(resid(Y.lm))
qqline(resid(Y.lm),col="blue")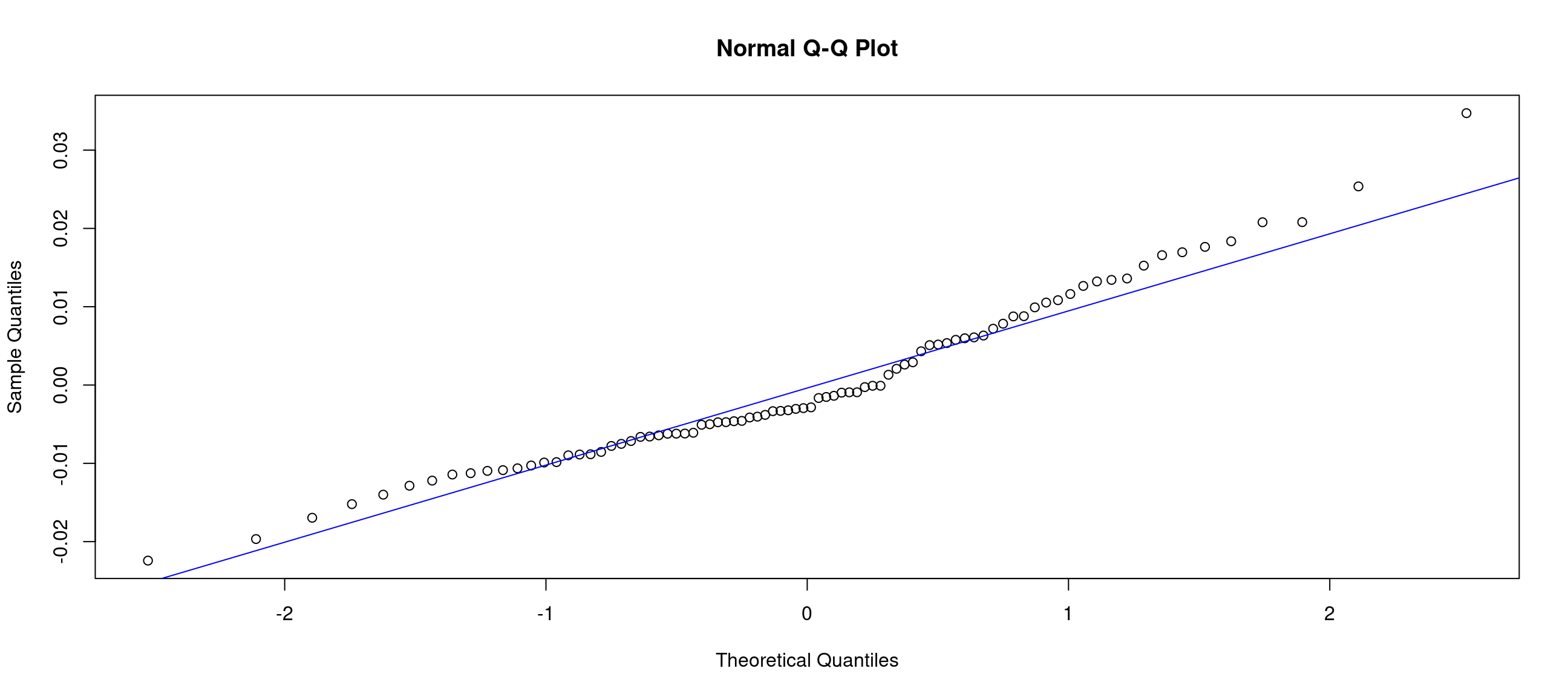
L’analyse de la variance
L’analyse de variance est une méthode statistique permettant de comparer des groupes. Elle peut être utilisée, par exemple, pour rechercher l’action d’un facteur sur une variable d’intérêt. Nous illustrerons cette méthode avec le jeu de données “porcelets” relatif à l’augmentation du poids de i porcelets, i ∈ {1, . . . , n}, en fonction du type d’alimentation de leur mère. Dans cette expérience, trois types d’aliments sont utilisés. Ils correspondent aux niveaux du facteur aliment :
levels(porcelets$aliment)NULLOn peut représenter les données sous forme de boxplots pour chacun des groupes j, j ∈ {al1, al2, T } (Figure 17). Dans la suite, nous noterons x ij l’observation du porcelet i dont la mère reçoit l’aliment j. Pour savoir s’il existe une influence de l’alimentation de la mère sur la croissance des porcelets, il faut comparer la part de variabilité due à l’alimentation de la mère et la part de variabilité résiduelle (variabilité entre les porcelets d’un même groupe). Pour cela, on calcule la somme des carrés des écarts à la moyenne intra groupes (x ij − x .j ) 2 que l’on notera “SSW”, et la somme des carrés des écarts à la moyenne inter groupes (x .j − x .. ) 2 que l’on notera “SSB”. La somme des carrés totale (x ij − x .. ) 2 , notée “SST”, peut ainsi se décomposer :
SST = SSW + SSB
La fonction aov() calcule les sommes de carrés intra et inter groupes.
pds.aov <- aov(pds ~ aliment, data=porcelets)
pds.aovCall:
aov(formula = pds ~ aliment, data = porcelets)
Terms:
aliment Residuals
Sum of Squares 35.41750 54.10875
Deg. of Freedom 2 21
Residual standard error: 1.605181
Estimated effects may be unbalancedChaque somme de carrés est associée à un degré de liberté (df) qui est pour l’aliment, son nombre de niveaux - 1, et pour la résiduelle, le nombre total d’observations - le df de l’aliment -1.
La fonction anova() réalise les tests statistiques à partir des résultats de l’aov et produit un tableau d’analyse de variance.
anova(pds.aov)Analysis of Variance Table
Response: pds
Df Sum Sq Mean Sq F value Pr(>F)
aliment 2 35.418 17.7088 6.8729 0.005056 **
Residuals 21 54.109 2.5766
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Ce tableau d’analyse de variance montre que la probabilité associée à l’effet de l’alimentation de la mère sur la croissance des porcelets est significative (si on considère un seuil de significativité à 0.05). Cela veut dire qu’au moins une des différences entre les 3 groupes est significative. On peut en conclure que, globalement, l’alimentation de la mère influe sur la croissance des porcelets. On peut aussi vouloir comparer les groupes 2 à 2. Pour cela, on utilise la fonction pairwise.t.test() qui réalise des comparaisons par paires basées sur des tests de Student. Une option de cette fonction permet de spécifier la méthode d’ajustement, en cas de comparaisons multiples. Par défault, cette option est “holm”.
pairwise.t.test(porcelets$pds,porcelets$aliment,p.adjust.method="bonferroni")
Pairwise comparisons using t tests with pooled SD
data: porcelets$pds and porcelets$aliment
al1 al2
al2 0.1041 -
T 0.5132 0.0042
P value adjustment method: bonferroni pairwise.t.test(porcelets$pds,porcelets$aliment,p.adjust.method="fdr")
Pairwise comparisons using t tests with pooled SD
data: porcelets$pds and porcelets$aliment
al1 al2
al2 0.0520 -
T 0.1711 0.0042
P value adjustment method: fdr Les résultats affichés montrent dans les 2 cas une différence significative de la croissance des porcelets entre les groupes T et al2. La différence entre les groupes al1 et al2 n’apparaı̂t pas significative si la méthode d’ajustement choisie est celle de Bonferonni.
La fonction t.test() est plus générale dans le sens où elle permet de faire un test de Student pour comparer deux groupes (vecteurs) de façon appariée ou non (par défaut) avec variance égale ou non (par défaut).
Par exemple :
t.test(porcelets$pds[porcelets$aliment== 'T' ],porcelets$pds[porcelets$aliment== 'al2' ])
Welch Two Sample t-test
data: porcelets$pds[porcelets$aliment == "T"] and porcelets$pds[porcelets$aliment == "al2"]
t = 3.6747, df = 13.473, p-value = 0.002651
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
1.221853 4.678147
sample estimates:
mean of x mean of y
7.075 4.125 De plus la fonction p.adjust() permet d’ajuster un ensemble de p-values pour tenir compte d’une comparaison multiple.
intercept <- Y.lm$coeff[1]
pente <- Y.lm$coeff[2]
plot(Y$gtg,Y$aaa)
abline(a=intercept, b=pente, col="blue")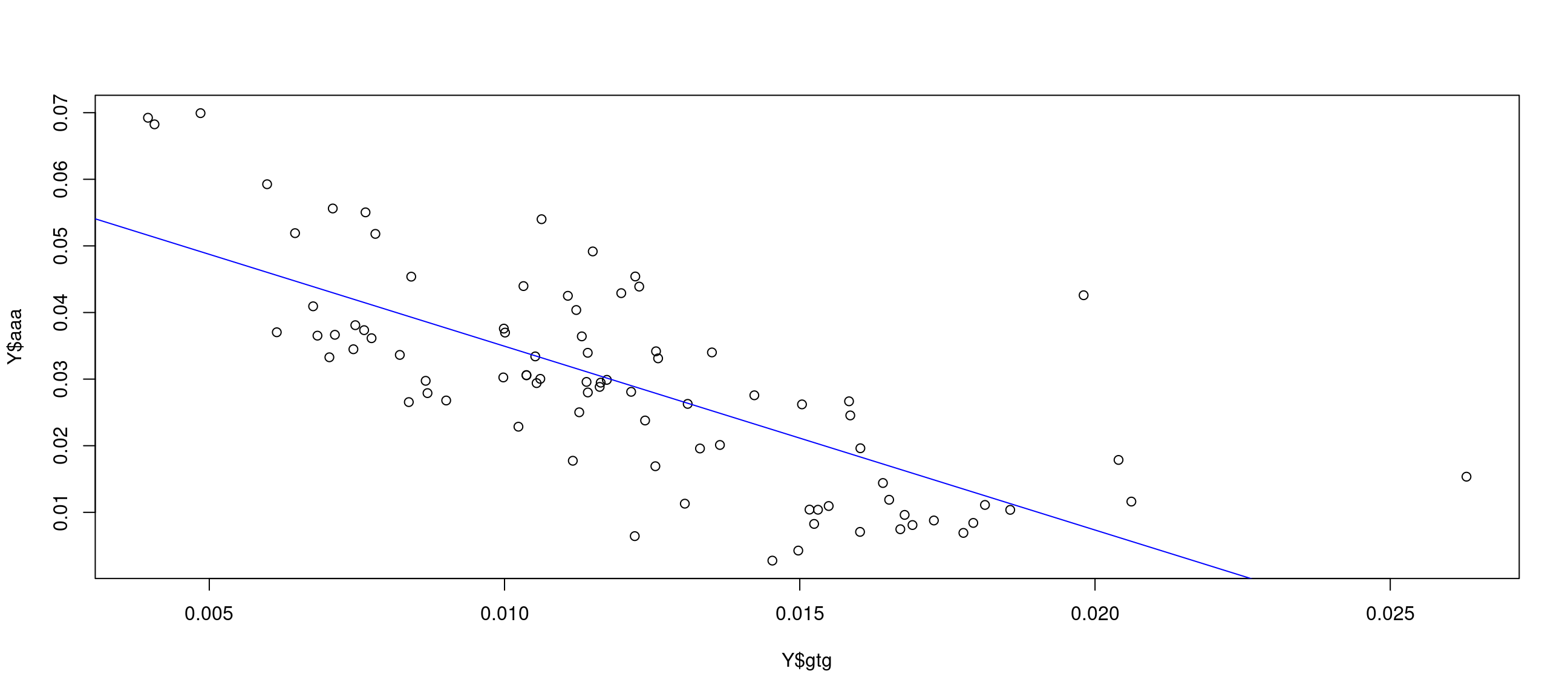
plot(porcelets$pds)